Ero sivun ”Massadata kansanterveyden edistämisessä” versioiden välillä
(→Suomalaisia massadatan esimerkkejä) |
(→Johdanto) |
||
| (Yhtä välissä olevaa versiota samalta käyttäjältä ei näytetä) | |||
| Rivi 20: | Rivi 20: | ||
{{defend|# |Kaikki viitteet tiedostossa BigData.zip pitäisi kirjoittaa <nowiki><ref>-muotoon.</nowiki> VIitteen nimeksi tulee ensimmäisen kirjoittajan sukunimi pienellä kirjoitettuna. Jos on monta artikkelia yhdellä kirjoittajalla, laitetaan vuosiluku perään ilman välejä, sitten a, b, c jne.|--[[Käyttäjä:Jouni|Jouni Tuomisto]] ([[Keskustelu käyttäjästä:Jouni|keskustelu]]) 31. toukokuuta 2015 kello 10.14 (UTC)}} | {{defend|# |Kaikki viitteet tiedostossa BigData.zip pitäisi kirjoittaa <nowiki><ref>-muotoon.</nowiki> VIitteen nimeksi tulee ensimmäisen kirjoittajan sukunimi pienellä kirjoitettuna. Jos on monta artikkelia yhdellä kirjoittajalla, laitetaan vuosiluku perään ilman välejä, sitten a, b, c jne.|--[[Käyttäjä:Jouni|Jouni Tuomisto]] ([[Keskustelu käyttäjästä:Jouni|keskustelu]]) 31. toukokuuta 2015 kello 10.14 (UTC)}} | ||
| + | <ref name="altaf-ul-amin">Md. Altaf-Ul-Amin, Farit Mochamad Afendi, Samuel Kuria Kiboi, and Shigehiko Kanaya; [http://www.hindawi.com/journals/bmri/2014/428570/ Systems Biology in the Context of Big Data and Networks]</ref> | ||
| + | |||
| + | <ref name="bolouri">Hamid Bolouri; [http://www.cell.com/trends/genetics/abstract/S0168-9525%2814%2900030-4?_returnURL=http%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS0168952514000304%3Fshowall%3Dtrue Modeling genomic regulatory networks with big data]</ref> | ||
| + | |||
| + | <ref name="choudhury">Suparna Choudhury, Jennifer R. Fishman , Michelle L. McGowan and Eric T. Juengst; Big data, open science and the brain: lessons learned from genomics. doi: 10.3389/fnhum.2014.00239</ref> | ||
| + | |||
| + | <ref name="chute">Christopher G. Chute, MD, DrPH, Mollie Ullman-Cullere, MS, MSE, Grant M. Wood, BS, Simon M. Lin, MD, Min He, PhD, and Jyotishman Pathak, PhD; Some experiences and opportunities for big data in translational research. doi:10.1038/gim.2013.121.</ref> | ||
| + | |||
| + | <ref name="cook">J. A. Cook and G. S. Collins; The rise of big clinical databases. DOI: 10.1002/bjs.9723</ref> | ||
| + | |||
| + | <ref name="cunningham">John P Cunningham & Byron M Yu; Dimensionality reduction for large-scale neural recordings. [http://www.nature.com/neuro/journal/v17/n11/full/nn.3776.html doi:10.1038/nn.3776]</ref> | ||
| + | |||
| + | <ref name="dai">Lin Dai, Xin Gao, Yan Guo, Jingfa Xiao and Zhang Zhang; [http://www.biologydirect.com/content/7/1/43 Bioinformatics clouds for big data manipulation]</ref> | ||
| + | |||
| + | <ref name="dilisizian">Steven E. Dilsizian & Eliot L. Siegel; Artificial Intelligence in Medicine and Cardiac Imaging: Harnessing Big Data and Advanced Computing to Provide Personalized Medical Diagnosis and Treatment. DOI 10.1007/s11886-013-0441-8</ref> | ||
| + | |||
| + | <ref name="dong">Dong et al. Reverse enGENEering of Regulatory Networks from Big Data: A Roadmap for Biologists. Bioinformatics and Biology Insights 2015:9 61–74 doi: 10.4137/BBI.S12467.</ref> | ||
| + | |||
| + | <ref name="editorial">Focus on big data. Nature Neuroscience editorial, Volume 17, number 11, November 2014</ref> | ||
| + | |||
| + | <ref name="ferguson">Adam R Ferguson, Jessica L Nielson, Melissa H Cragin, Anita E Bandrowski & Maryann E Martone; Big data from small data: data-sharing in the ‘long tail’ of neuroscience. Nature Neuroscience, Volume 17, number 11, November 2014</ref> | ||
| + | |||
| + | <ref name="gomez-martin">Alex Gomez-Marin, Joseph J Paton, Adam R Kampff, Rui M Costa & Zachary F Mainen; Big behavioral data: psychology, ethology and the foundations of neuroscience. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3812</ref> | ||
| + | |||
| + | <ref name="greene">Casey S. Greene, Jie Tan, Matthew Ung, Jason H. Moore and Chao Cheng; Big Data Bioinformatics. J. Cell. Physiol. 229: 1896–1900, 2014. DOI: 10.1002/jcp.24662</ref> | ||
| + | |||
| + | <ref name="kao">Rowland R. Kao, Daniel T. Haydon, Samantha J. Lycett, and Pablo R. Murcia; Omics: Fulfilling the Promise. [http://www.cell.com/trends/microbiology/abstract/S0966-842X%2814%2900046-8?_returnURL=http%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS0966842X14000468%3Fshowall%3Dtrue Supersize me: how whole-genome sequencing and big data are transforming epidemiology]</ref> | ||
| + | |||
| + | <ref name="kitchen">Robert R Kitchen, Joel S Rozowsky, Mark B Gerstein & Angus C Nairn; Decoding neuroproteomics: integrating the genome, translatome and functional anatomy. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3829</ref> | ||
| + | |||
| + | <ref name="lichtman">Jeff W Lichtman, Hanspeter Pfister & Nir Shavit; The big data challenges of connectomics. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3829</ref> | ||
| + | |||
| + | <ref name="maze">Ian Maze, Li Shen, Bin Zhang, Benjamin A Garcia, Ningyi Shao, Amanda Mitchell, HaoSheng Sun, Schahram Akbarian, C David Allis & Eric J Nestler; Analytical tools and current challenges in the modern era of neuroepigenomics. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3829</ref> | ||
| + | |||
| + | <ref name="merelli">Ivan Merelli, Horacio Pérez-Sánchez, Sandra Gesing, and Daniele D’Agostino; [http://www.hindawi.com/journals/bmri/2014/134023/ Managing, Analysing, and Integrating Big Data in Medical Bioinformatics: Open Problems and Future Perspectives], BioMed Research International. Volume 2014, Article ID 134023</ref> | ||
| + | |||
| + | <ref name="poldrack">Russell A Poldrack & Krzysztof J Gorgolewski; Making big data open: data sharing in neuroimaging. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3829</ref> | ||
| + | |||
| + | <ref name="sejnowskia">Terrence J Sejnowski, Patricia S Churchland, and J Anthony Movshon; Putting big data to good use in neuroscience. Published as: Nat Neurosci. 2014 November ; 17(11): 1440–1441.</ref> | ||
| + | |||
| + | <ref name="sejnowskib>Terrence J Sejnowski, Patricia S Churchland & J Anthony Movshon; Putting big data to good use in neuroscience. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3829</ref> | ||
| + | |||
| + | <ref name="shin">Jaehoon Shin, Guo-li Ming & Hongjun Song; Decoding neural transcriptomes and epigenomes via high-throughput sequencing. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3829</ref> | ||
| + | |||
| + | <ref name="wang">Weiqi Wang, PhD, Eswar Krishnan, MD, MPH; [http://www.medinform.jmir.org/2014/1/e1/ Big Data and Clinicians: A Review on the State of the Science.]</ref> | ||
| + | |||
| + | <ref name="wu">Hongyan Wu, Atsuko Yamaguchi; Semantic Web technologies for the big data in life sciences. BioScience Trends. 2014; 8(4):192-201.</ref> | ||
| + | |||
| + | <ref name="yoo">Changwon Yoo, Luis Ramirez, Juan Liuzzi; [http://dx.doi.org/10.5213/inj.2014.18.2.50 Big Data Analysis Using Modern Statistical and Machine Learning Methods in Medicine]. http://dx.doi.org/10.5213/inj.2014.18.2.50</ref> | ||
| + | |||
| + | <ref name="zou">Dong Zou, Lina Ma, Jun Yu, Zhang Zhang; [http://dx.doi.org/10.1016/j.gpb.2015.01.006 Biological Databases for Human Research.] Genomics Proteomics Bioinformatics 13 (2015) 55–63</ref> | ||
Artikkelissa esitetään katsaus joihinkin massadatan keskeisiin ja ajankohtaisiin piirteisiin erityisesti kansanterveyden, lääketieteen ja yhteiskunnallisen päätöksenteon kannalta. Katsauksessa ei siis pyritä tämän laajan aihepiirin kattamiseen vaan kiinnostavien kysymysten esiinnostamiseen. | Artikkelissa esitetään katsaus joihinkin massadatan keskeisiin ja ajankohtaisiin piirteisiin erityisesti kansanterveyden, lääketieteen ja yhteiskunnallisen päätöksenteon kannalta. Katsauksessa ei siis pyritä tämän laajan aihepiirin kattamiseen vaan kiinnostavien kysymysten esiinnostamiseen. | ||
| Rivi 57: | Rivi 108: | ||
| Monitieteinen ala, jossa laskennallisin menetelmin tutkitaan biologisia tietoaineistoja... | | Monitieteinen ala, jossa laskennallisin menetelmin tutkitaan biologisia tietoaineistoja... | ||
|} | |} | ||
| − | |||
=== Lääketieteeseen liittyviä tietokantoja === | === Lääketieteeseen liittyviä tietokantoja === | ||
| Rivi 416: | Rivi 466: | ||
=== Viitteet === | === Viitteet === | ||
| − | <references | + | </references> |
* ''Kirjallisuusviitteiden esittämisessä noudatetaan Vancouver-järjestelmää (tekstiin numeroidut viitteet) ja kirjallisuusluettelo laaditaan lehden yleisten kirjoitusohjeiden mukaan, esim. Dagfinrud H, Kvien TK, Hagen KB. Physiotherapy interventions for ankylosing spondylitis. The Cochrane Database of Systematic Rewievs 2013, Issue 4. Art. No.:CD002822. DOI: 10.1002/14651858.CD2822.pub2. Teemanumeron katsausta ei tulisi kuormittaa viitteillä (max. 30). | * ''Kirjallisuusviitteiden esittämisessä noudatetaan Vancouver-järjestelmää (tekstiin numeroidut viitteet) ja kirjallisuusluettelo laaditaan lehden yleisten kirjoitusohjeiden mukaan, esim. Dagfinrud H, Kvien TK, Hagen KB. Physiotherapy interventions for ankylosing spondylitis. The Cochrane Database of Systematic Rewievs 2013, Issue 4. Art. No.:CD002822. DOI: 10.1002/14651858.CD2822.pub2. Teemanumeron katsausta ei tulisi kuormittaa viitteillä (max. 30). | ||
Versio 1. kesäkuuta 2015 kello 13.25
Massadata kansanterveyden edistämisessä on katsausartikkeli Big Datasta. Se on kirjoitettu Duodecim-lehden teemanumeroon.
Kirjoitusohjeita (ks. myös N:\YMAL\Publications\In_progress\Tuomisto_Duodecim):
- Valistuneelle kohdeyleisölle, joka ei kuitenkaan ole alan asiantuntija.
- DL 15.5.2015 Eija Rautiaiselle ((at)duodecim.fi)
- Teemanumeron katsauksen pituus saa olla enintään 1 800 sanaa + kuvat + taulukot.
- Artikkelissa tulee olla otsikkosivu, jossa on tekijöiden nimet, oppiarvot ja virka-asemat sekä laitosten nimet. Yhteyskirjoittajan osalta mainitaan lisäksi postiosoite.
- Katsausten väliotsikoinnissa käytetään kaksiportaista asteikkoa (väliotsikko ja rivinalkuinen kursivoitu alaotsikko).
- Kuvat tulisi lähettää erillistiedostoina, mielellään giff- tai jpg -muodossa.
- Katsausartikkelin tehtävä on kertoa lukijalle käsiteltävästä aiheesta moniulotteisesti, asiantuntevasti ja tasapainoisesti. Oppikirjamaisuutta tulee välttää ja katsauksen onkin oltava kriittinen ja tutkimushavaintoja kypsästi tulkitseva. Pelkkä tutkimusten referointi ei riitä, vaan kirjoittajan on jäsennettävä tutkimusten tuloksia mahdollisimman laajoihin yhteyksiin. Aiheen tulee kiinnostaa suurinta osaa lehden lukijoita, joten myös kirjoituksen tyylin pitää tukea yleistä ymmärrettävyyttä. Katsauksessa olisi kyettävä – aiheen mukaan – tarjoamaan tietoa ja tukea myös niihin valintoihin, joita käytännön lääkäri joutuu arkityössään tekemään, kuten hoidon porrastukseen.
Sisällysluettelo
- 1 Tiivistelmä
- 2 Johdanto
- 3 Lääketieteeseen liittyviä tietokantoja
- 4 Suomalaisia massadatan esimerkkejä
- 5 Massadatan käyttökohteita
- 6 Massadatan menetelmäkokonaisuus
- 7 Datan analysointi- ja hyödyntämismenetelmiä
- 8 Datan jakaminen
- 9 Massadatan haasteet
- 10 Lopuksi
- 11 Ydinasiat
- 12 Avainsanat
- 13 Viitteet
Tiivistelmä
- Tiivistelmä artikkelin sisällöstä suomeksi ja englanniksi. Maksimipituus 100 sanaa.
Johdanto
←--#: . Kaikki viitteet tiedostossa BigData.zip pitäisi kirjoittaa <ref>-muotoon. VIitteen nimeksi tulee ensimmäisen kirjoittajan sukunimi pienellä kirjoitettuna. Jos on monta artikkelia yhdellä kirjoittajalla, laitetaan vuosiluku perään ilman välejä, sitten a, b, c jne. --Jouni Tuomisto (keskustelu) 31. toukokuuta 2015 kello 10.14 (UTC) (type: truth; paradigms: science: defence)
Artikkelissa esitetään katsaus joihinkin massadatan keskeisiin ja ajankohtaisiin piirteisiin erityisesti kansanterveyden, lääketieteen ja yhteiskunnallisen päätöksenteon kannalta. Katsauksessa ei siis pyritä tämän laajan aihepiirin kattamiseen vaan kiinnostavien kysymysten esiinnostamiseen.
Massadatan käyttö on edennyt lääketieteen alueella hitaammin kuin monilla muilla alueilla (mm. Dilsizian) [27]
Neuromittausdatan avaaminen on ollut hidasta Choudbury
- Mitä on big data eli massadata?
- Muita olennaisia termejä (taulukko 1)
| Termi | Selite |
|---|---|
| Massadata | Englanniksi big data. Suuret data-aineistot, joiden tilastollinen analyysi ei tahdo onnistua tavanomaisin menetelmin. |
| Avoin data | Englanniksi open data tai open linked data. Tietoa, joka on saatavissa koneluettavassa muodossa maksutta ja niin, että tekijänoikeudet eivät rajoita sen jatkokäyttöä. |
| Omadata | Englanniksi MyData. Henkilöä itseään koskeva data, jonka käytöstä hän voi itse päättää. Tällä hetkellä ollaan rakentamassa käytäntöjä, joiden avulla henkilö voi antaa tai evätä tietojensa käyttöluvan haluamilleen tahoille omadataoperaattorinsa välityksellä riippumatta siitä, missä ja kenen järjestelmissä tieto sijaitsee. Henkilö voi myös lukea ja joissain tapauksissa korjata tai täydentää omadataansa. |
| Keinodata | Avoin data, joka ulkomuodoltaan ja tilastollisilta ominaisuuksiltaan muistuttaa jotakin arkaluontoista dataa kuten potilasaineistoa ilman, että siitä voisi kuitenkaan päätellä kenenkään todellisen henkilön tietoja. Keinodataa voi käyttää tutkimuskysymysten ja tilastoanalyysien muotoilemiseen ja alustavaan testaamiseen, ennen kuin analyysi ajetaan vastaavalla todellisella datalla. |
| Tietokide eli avoin muuttuja | Englanniksi open variable tai info crystal. Koneluettavassa muodossa oleva vastaus johonkin täsmälliseen tutkimuskysymykseen. Tietokide on netissä jatkuvasti päivittyvä ja kiteytyvä kuvaus kaikesta siitä tiedosta, joka kyseisestä kysymyksestä on ehditty kerätä, samaan tapaan kuin Wikipedia-artikkeli. Tietokide sisältää myös ymmärrettävät ja uskottavat perustelut sille, miten havaintodata ja muu tieteellinen tieto on jalostettu vastaukseksi kysymykseen. Tyypillisesti avoimen muuttujan vastaus on ehdollinen todennäköisyysjakauma ja sisältää tietoa myös niistä kausaalisista ja muista tekijöistä, jotka voivat vaikuttaa vastaukseen. |
| Datan pitkä häntä (taustadata) | Englanniksi long-tail data. Tarkoittaa laajaa joukkoa pieniä aineistoja, jotka kokonsa tai merkityksensä puolesta jäävät myös helposti julkaisematta. Näitä ovat esimerkiksi erilliset tietokannat, nollatulokset ja eläinkoepäiväkirjat. Ferguson nn3838 LUE! |
| Bioinformatiikka | Monitieteinen ala, jossa laskennallisin menetelmin tutkitaan biologisia tietoaineistoja... |
Lääketieteeseen liittyviä tietokantoja
- Haasteita ja mahdollisuuksia kansanterveyden tutkimuksessa.
- Suomalaisten tietoaineistojen mahdollisuudet.
- Henkilötietojen suojaamisen haasteet.
- Keinodata eli artificial data, katso op_en:ReplicaX.
- Omadata eli MyData
Terveysdatan kannalta tärkeitä massadatan tietokantoja.
- World map, KAMPO, JAMU, Tea Pot (Altaf-Ul-Amin et al)
Zou: Erityyppiset tietokannat. Esitellään eri tyypit ja ehkä mainitaan esimerkkejä mutta muuten viitataan artikkeliin. DNA, RNA; proteiini, ...
Cook: Large clinical database research studies are becoming ubiquitous. This is to be welcomed, with potential benefits including representativeness, data from large numbers of patients, identification of systemic problems, and precision of estimation in statistical analyses. They are particularly well suited to summarizing a population, and looking at associations and variations between groups or over time. However, the limitations of such data sources must not be overlooked, and each research study needs to be considered carefully in its own right and the validity of the data used for this purpose. Large numbers should not preclude or supersede assessment of the relevance, fitness for purpose and reliability of the data collected, and the statistical methodology adopted.
Cook:
Table 1 Types of clinical database and examples
Administrative databases
Routine data collected for the purpose of administering healthcare. The data can be used to monitor activity and support local service planning as well as for some form of clinical quality assessment. An important distinction is between where the data are being collected primarily for clinical management and process monitoring purposes, and where they are used for health insurance reimbursement. Only limited patient characteristics (e.g. age, sex, postcode and presentation) along with diagnosis and procedure information are typically recorded
Clinical management database example: The Hospital Episode Statistics (HES)12 database is an administrative database of hospital admission, outpatient appointments, and accident and emergency attendances at National Health Service hospitals in England. Data collection ends once the patient has completed the hospital episode (e.g. discharged from hospital)
Health insurance reimbursement claims database example: The Medicare Provider Analysis and Review file (MedPAR)13 is an administrative database that contains data on Medicare beneficiaries who receive services at inpatient facilities in the USA. Each Medicare beneficiary has a unique identification number
Clinical databases
These include databases in which the data have been collected specifically to assess a particular clinical outcome. Eligibility may be disease-based or procedure/device-based. In contrast to administrative data, important relevant clinical factors are collected, and also a wider range of outcomes and information on co-morbidities
Disease-based clinical database example: The Swedish Colon Cancer Registry2, set up in 2007, collects data on all patients diagnosed with adenocarcinoma of the colon in Sweden. The primary aim of this registry is to monitor clinical quality. Data collected include patient demographics and diagnosis, preoperative management, surgical procedure, pathology report and outcomes.
Treatment-based clinical database example: The American College of Surgeons National Surgical Quality Improvement Program (ACS NSQIP) database collects data on patients undergoing major surgery at a participating institution; these are mostly US Department of Veteran Affairs hospitals, although there are some private institutions and hospitals in other countries. The data collection was set up specifically to enable risk-adjusted clinical outcome to be calculated. Data are collected on a sample of eligible patients for each institution, and include patient demographics, preoperative medical history, intraoperative data, clinical findings (e.g. postoperative diagnosis) and laboratory investigations, along with postdischarge outcome data (complications, further procedures and mortality) for 30 days after surgery
Suomalaisia massadatan esimerkkejä
Taulukkona: Lyhyt kuvaus, kommentti onko avoimesti saatavissa ja nettilinkki seuraavista
- HILMO hoitoilmoitusrekisteri
- Avohilmo avoterveydenhuollon hoitoilmoitusrekisteri
- Rokotusrekisteri
- Yhti ympäristöterveydenhuollon tietokanta
- Kuti Elintarviketutvallisuustietokanta
- Hertta Ympäristötietokanta
- Syöpärekisteri
Massadata-artikkeli Omadataraporttia referoitava Löytyykö suomalaisia massadatastrategioita Kommentoitava evidence-based suuntausta Suomessa Etsi viite replicaXstä. Onko peräti julkaisua? Massadatasta kannattaa erikseen mainita ihmisten itsensä keräämät terveys oire ja kuntoilutiedot, jotka hyvinkin poan saattavat olla hoitavan lääkärin käytettävissä. Miten niitä sitten käytettäisiin? Suomalaisiin tietokantaeImerkkeihin myös ylläpitäjä Opiskeltava Suomen big data strategia Sote-tieto hyötykäyttöön myös kuvattava Samoin avoiment tiedon ovjelma ja avoimen tieteen ja tutkimuksen strategia sekä ATT Haaste: lock-in ja toimittajariipouvuus. Suomi voisi tarjota ilmaisia peerusratkaisuja käyttöön ja saisi näin datat jalostettavaksi. Vrt Apotti (kannattaako mainita?)
Big data strategia:
Suomem edut: perusrekisterit, osaaminen ja i fra ovat hyvät. Massadata korostaa yhteistyön ja avoimuuden merkitystä. Monialaisuus ja täydennyskoulutus tarpeen edistämisessä. Teknologia analyysimenetelmät ja substanssitieto saatava kohtaamaan. Fimmin geenitiedot? Suomessa olisi innovaatiotilausta erityisesti ikääntyvälle väestönosalle suunnatuille ennaltaehkäisevän terveydenhuollon ja itsehoidon palveluille. OECD:n Suo- mea koskevan raportin mukaan40 väestön ikääntymisen aiheuttamat rakenteelliset ter- veydenhuollon kustannuspaineet tulevat lähivuosina ja myös tulevaisuudessa olemaan Suomessa merkittävämpi ongelma kuin useimmissa muissa OECD-maissa.
Suomessa on alan osaamista esimerkiksi bioinformatiikan ja molekyylilääketieteen alalla. Toinen tärkeä alue liittyy kansalaisten omatoimiseen terveydenhoitoon ja hyvinvoinvointiin liittyvien itsemit- tausteknologioiden kehitykseen, jossa suomalaisyrityksissä on osaamista kymmenien vuosien ajalta.
Big datalta odotetaan terveydenhuollon laadun parantumista ja säästöjä. Data-analyysin odotetaan kehittyvän tavalla, joka auttaa yksittäisen kansalaisen terveysriskien ennus-
tamisessa erilaisten datavirtojen avulla sekä sairauksien ehkäi- syssä. Terveystietojen hyöty- käyttöön liittyy kuitenkin yksi- tyisyyteen, sosiaalisiin ja eetti- siin näkökulmiin liittyviä kysy-
”Suuret tietoaineistot parantavat omahoitoa, ennaltaehkäisevää toimintaa, valvontaa sekä tukevat ammattilaisten työtä”
Soteuudistus tarjoaa merkittävän mahdollisuuden
Omahoitomenetelmät ja suomalai en mittausteknologia luovat mahdollisuuksia.
Esimerkkejä:
Syöpälääkkeiden vaikutusta soluihin tutkitaan genominlaajuisilla matemaattisilla malleilla.
Lääkeaineiden tehon vaikutus soluissa ennustetaan matemaattisin mallein koneoppimisen avulla.
Muun muassa eri- laisen mobiilidatan, trendidatan sekä sosiaalisesta mediasta saatavan datan määrän kas- vu tuo tutkijoiden ulottuville aivan uudenlaisia aineistoja. Erimerkiksi ihmisten todellista käyttäytymistä kuvaavat verkonkäyttö- ja paikkatietoaineistot mahdollistavat merkittävi- en lisätietojen saamisen ja siten täydentävät (tai jopa korvaavat) perinteisiä kyselytutki- muksia, joilla tutkitaan muun muassa ihmisten käyttäytymisaikomuksia.
Massadatan käyttökohteita
Tässä vain nostetaan joitakin esimerkkejä
Here, we examine three important motivations for population studies: single-trial hypotheses requiring statistical power, hypotheses of population response structure and exploratory analyses of large data sets. Methods to reduce dimensionality in data. Cunningham
Cook: Common uses of the data include: providing population characteristics; identifying risk factors and developing prediction (diagnostic or prognostic) models; observational studies comparing different interventions; exploring variation between healthcare providers; and as a supplementary source of data for another study. The main advantages of using such big data sources are their comprehensive nature, the relatively large number of patients they comprise, and the ability to compare healthcare providers. The main challenges are demonstrating data quality and confidently applying a causal interpretation to the study findings.
Massadatan menetelmäkokonaisuus
Dai: Illustration of bioinformatics cloud.
Data as a Service
- Public datasets
- Biological databases
Software as a Service
- Tools
- Pipelines
Platforms as a Service
- Analysis platforms
- Programming environments
Infrasctructures as a Service
- Virtual machines
- Virtualised resources
- Table 1 Cloud resources in bioinformatics (Dai et al)
Resource Description & availability Data as a Service (DaaS): AWS Public Datasets Cloud-based archives of GenBank, Ensembl, 1000 Genomes, Model Organism Encyclopedia of DNA Elements, Unigene, Influenza Virus, etc.; http://aws.amazon.com/publicdatasets Software as a Service (SaaS): BGI Cloud (unpublished) Cloud-based implementations of various genomic analysis applications; http://cloud.genomics.cn CloudAligner [16] Fast and full-featured MapReduce-based tool for sequence mapping; http://cloudaligner.sourceforge.net CloudBLAST [19] A cloud-based implementation of NCBI BLAST; http://ammatsun.acis.ufl.edu/amwiki/index.php/CloudBLAST_Project CloudBurst [17] Highly sensitive short read mapping with MapReduce; http://cloudburst-bio.sourceforge.net Contrail (unpublished) Cloud-based de novo assembly of large genomes; http://contrail-bio.sourceforge.net Crossbow [18] Read Mapping and SNP calling using cloud computing; http://bowtie-bio.sf.net/crossbow EasyGenomics (unpublished) Cloud-based NGS pipelines for whole genome resequencing, exome resequencing, RNA-Seq, small RNA and de novo assembly; http://www.easygenomics.org eCEO [26] Cloud-based identification of large-scale epistatic interactions in genome-wide association study (GWAS); http://www.comp. nus.edu.sg/~wangzk/eCEO.html FX [20] RNA-Seq analysis tool; http://fx.gmi.ac.kr Gaea (unpublished) Cloud-based genome re-sequencing assembly; http://bgiamericas.com/data-analysis/cloud-computing Hecate (unpublished) Cloud-based de novo assembly; http://bgiamericas.com/data-analysis/cloud-computing Jnomics (unpublished) Cloud-scale sequence analysis suite based on Apache Hadoop; http://sourceforge.net/apps/mediawiki/jnomics Myrna [21] Differential gene expression tool for RNA-Seq; http://bowtie-bio.sourceforge.net/myrna PeakRanger [24] Cloud-enabled peak caller for ChIP-seq data; http://www.modencode.org/software/ranger RSD [23] Reciprocal smallest distance algorithm for ortholog detection using Amazon's Elastic Computing Cloud; http://roundup.hms. harvard.edu VAT [25] Variant annotation tool to functionally annotate variants from multiple personal genomes at the transcript level; http://vat. gersteinlab.org YunBe [22] Pathway-based or gene set analysis of expression data; http://tinyurl.com/yunbedownload Platform as a Service (PaaS): Eoulsan [27] Cloud-based platform for high throughput sequencing analyses; http://transcriptome.ens.fr/eoulsan Galaxy Cloud [28,29] Cloud-scale Galaxy for large-scale data analysis; http://galaxy.psu.edu Infrastructure as a Service (IaaS): Cloud BioLinux [30] A publicly accessible virtual machine for high performance bioinformatics computing using cloud platforms; http:// cloudbiolinux.org CloVR [31] A portable virtual machine for automated sequence analysis using cloud computing; http://clovr.org
Datan analysointi- ja hyödyntämismenetelmiä
Bayes-verkot Yoo LUE!
Gene regulatory networks Bolouri; metodipaperi
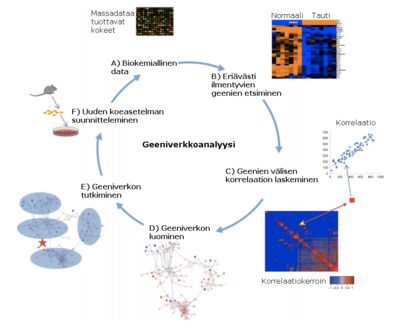
Network analysis Omics data -> Find DEGs -> Calculate partial correlation between DEGs -> Network building -> Network interrogation -> Design new experiments to test hypotheses. (Dong et al)
Next generation sequencing. In less than a decade, NGS has changed the way and scale at which biology is studied. Many new tools are now available to characterize cells at the genome-wide level, including their DNA modifications, histone modifications, transcription factor binding, chromatin 3D interactions, accessible chromatin, the RNA transcriptome and many more. More and more new assays are becoming available, and NGS itself is rapidly evolving. Shin et al
Yhdennetyt genomiset, transkriptomiset ja proteomiset analyysimenetelmät. Kitchen: Box 1 An ideal integrated multi-omic analysis Such an analysis would exploit information garnered at each stage of the gene-expression process to improve the overall utility of results obtained from RNA and protein profiling (Fig. 1). Information regarding genomic variants in a given sample or individual can be used to create a personalized genome for that individual. These homozygous and heterozygous variants are incorporated in transcriptomic analysis by RNA-seq and enable the detection of allelic imbalance (ASE), in addition to information relating to alternative splicing and RNA editing (RDDs). This cascade of information in a given sample aids in the analysis of mass-spectrometry spectra, in which peptides may be identified that support or refute the presence of nonsynonymous alternative-splicing, RDD and ASE events. From the relative abundances of these peptides, it is possible to compare isoform abundance and allelic imbalance with the values estimated at the RNA level, allowing inferences to be made based on the proteomic data, such as the presence or absence of a particular protein isoform.
Datan jakaminen
Datan jakamisen parhaat käytännöt Ferguson Box 2. Wang LUE!
Semantic web, ontologia, linked data Wu LUE! (Merelli; tekninen)
Datan jakamisen käytännöt ja kustannukset neuropuolella Poldrak
ReplicaX ja keinodata
Massadatan haasteet
Datan jakamisen haasteet. Arvostus Poldrak LUE!, Editorial. Sejnowski nn
Sejnowski nn. Big datan tärkeät aiheet tai haasteet. 1) Big dataa on runsaasti mutta se on hajanaista, tuottu useille menetelmillä ja eläinlajeilla ja vaikeasti standardoitavissa. 2) Asioita pitää oppia peilaamaan (käyttäytymis)vasteisiin, mittaaminen onkin muuttunut helpommaksi ja vähemmän invasiiviseksi. Mutta kausaalisuuksien löytäminen on haastavaa. 3) Tieto laboratorioiden välillä liikkuu yleensä kokousten ja artikkelien muodossa muuta dataa harvemmin jaetaan. 4) Datan kasvaessa sen analysointi vaikeutuu ja tarvitaan uusia algoritmejä. On myös opittava tuottamaan aineistoja, joista mielekkäiden yhteyksien löytäminen on mahdollista. 5) Nykyteoriat ovat selittäviä pikemmin kuin ennustavia ja perustuvat pieniin aineistoihin. Teorioista pitäisi tehdä selkeämpiä ja ennustavia vaikka se edellyttäisi tinkimistä matemaattisesta monimutkaisuudesta. Yleispäätelmä: tarvitaan kulttuurinmuutosta, matemaattisesti orientoituneita tutkijoita ja ennakkoluulottomia, kattavia ja testattavia teorioita.
Jotkin tekniikat tai aiheet ovat kalliita datan tuottamiseen, eikä sellaisia pysty käyttämään ellei ole tiedossa lähitulevaisuuden selkeä hyöty, jonka takia datantuottoon löytyy rahoittaja. Hankaluutena on, että usein hyödyt eivät ole etukäteen arvioitavissa. Konnektomiikka eli hermoston synapsirakenteen tutkiminen on esimerkki tällaisesta. Lichtman et al
Teoreettisten viitekehysten, koeasetelmien ja teknologisen kehityksen pitäisi kaikkien edistyä yhdessä tulosten saavuttamiseksi Gomez-Marin.
Tärkeä haaste on pystyä uuttamaan merkitystä ulos jatkuvasti lisääntyvästä datasta, ja tämä näkyy monilla aloilla kuten käyttäytymmistieteellisessä neurobiologiassa. Gomez-Marin.
Tietojen standardointi, tiedon uusiokäyttö ja syvempi yhteistyö kollegojen kanssa ovat tulevaisuuden suuntia. Gomez-Marin.
Potilastieto massadatana. Mining and advanced analysis of “big data” in health care provide the potential not only to perform “in silico” research but also to provide “real time” diagnostic and (potentially) therapeutic recommendations based on empirical data. “On demand” access to highperformance computing and large health care databases will support and sustain our ability to achieve personalized medicine. Dilsizian
Mahdollisuudet ja haasteen tekoälyn käytössä lääketieteessä Dilsizian
The combination of AI, big data, and massively parallel computing offers the potential to create a revolutionary way of practicing evidence-based, costeffective, and personalized medicine. However, barriers to adoption of AI technologies must be overcome from regulatory, legal, cultural, and political perspectives––even when technology solutions have matured. Dilsizian
Open access ja data sharing on välttämätöntä. Mitä haasteita ja oppeja on löydetty. Choudhury
Datan avaamisen haasteita Choudhury
- Tutkimuskulttuuri
- Muiden varastamat skuupit
- Miten jaetaan meriitti
- Tutkimuslupa annettu vain alkuperäiseen tarkoitukseen
- Potilas on vaarassa tunnistua kun kerätään paljon yksityiskohtaista dataa
- ym
Afurthersafeguardistohaveseparaterepositoriesfor summaryleveldataandindividualdata,thelatterofwhichis keptunderrestrictedaccess,asisspecifiedbyNIH’sGWASdata accesspolicy(Kaye, 2011; McEwenetal., 2013). Thesameistrue fortheHapMapprojectwhichusesCoriell’srepository,which has specificaccessrulesandregulations.Oftenaccesstodata is controlledbycommitteeswhomustdeterminewhetheruses ofthedataareappropriate,ethical,andfollowpolicyguidelines (McEwenetal., 2013). Choudhury
Aswehaveshown,thefieldofgenomicsdoesnotprovidea modelofstraightforwardsuccessindatasharingforbiomedical research.However,itsexperiencesandprecedencecanhelpthe neurosciencecommunityanticipatethechallengesandcomplex- itiesitislikelytoface. Choudhury
Information altruist: henkilö joka antaa oman biomedisiinisen datansa vapaaseen käyttöön (mm. Leena Palotie) Choudhury ←--#: . Etsi tietoa siitä, mitä ja miten Leena Palotie luovutti omaa dataansa tieteen käyttöön syövän tutkimiseksi --Jouni Tuomisto (keskustelu) 31. toukokuuta 2015 kello 20.51 (UTC) (type: truth; paradigms: science: defence)
The recent advent of next-generation sequencing technology allows the identification of trace- able differences in the pathogen genome that are trans- forming our ability to understand high-resolution disease transmission, sometimes even down to the host-to-host scale. Kao
High-throughput sequencing: the technological revolution that followed the Sanger sequencing technology that was used to generate the first complete human genome, allowing for mass generation of sequences at increasingly affordable costs. Currently broadly subdivided into next- or second-generation sequencing (Illumina or 454) and now third generation (PacBio). Kao
Bayesian model-based inference of infectious disease transmission. Kao
Individual level denominator data from at-risk populations are now being collected, often on a daily basis, and also increasingly in real time. Advances in computing power now provide the engine for mathematical and statistical techniques by which disparate datasets can be analyzed. Although we are only now arriving at this point, the combination of big data and tractable analytical techniques provides the opportunity to transform our approach to controlling infectious diseases in both epidemic and endemic contexts, with whole-genome sequencing (WGS) playing a leading role. Kao
Lopuksi
- Loppunousu, jossa ei pelkästään tiivistetä katsauksen viestiä vaan liitetään se laajempiin yhteyksiin tai pohditaan tulevaisuudennäkymiä.
Massadata mahdollistaa isot kulttuurimuutokset, mutta tiedolliset läpimurrot perustuvat pikemminkin näistä muutoksista johtuvaan ymmärryksen paranemiseen.
Tarve lisääntyy massadatasta jalostetuille kehittyneemmille tieto-olioille kuten tiedokeille (avoimille muuttujille).
Kahnemannin ja Tverskyn nopea ja hidas ajattelu nousee tärkeäksi osaksi massadataa, koska mahdollisuudet tuottaa verkossa yhteisöllisesti hidasta, rationaalista ajattelua paranevat eksponentiaalisesti avoimen datan ja massadatan ansiosta. Tällöin on mahdollista lisätä hitaan ajattelun merkitystä yhteiskunnallisten päätösten perustana.
Kliinikon näkökulmasta on tulossa ainakin kaksi muutosta: erilaiset henkilökohtaisen terveystiedon keräämisen välineet lisääntyvät ja potilas voi tarjota lääkärille esimerkiksi kännykän keräämää kuukausien liikuntadataa, jonka merkitystä hoidon kannalta pitäisi pystyä arvioimaan. Toisaalta potilaat pystyvät tekemään entistä perusteellisempia oma-arviointeja ja vertailuja netissä ja tarjota omia diagnoosi- ja hoitoehdotuksiaan lääkärille. Lääkärin rooli on muuttumassa asiantuntija-auktoriteetista valmentajaksi.
Ydinasiat
- Katsauksiin tulee laatia 3–5 erillistä virkettä, jotka lyhyesti tiivistävät kirjoituksen keskeisen sanoman. Virkkeet sijoitetaan Ydinasiat-tekstilaatikkoon.
Avainsanat
- 4-8 kpl
Viitteet
</references>
- Kirjallisuusviitteiden esittämisessä noudatetaan Vancouver-järjestelmää (tekstiin numeroidut viitteet) ja kirjallisuusluettelo laaditaan lehden yleisten kirjoitusohjeiden mukaan, esim. Dagfinrud H, Kvien TK, Hagen KB. Physiotherapy interventions for ankylosing spondylitis. The Cochrane Database of Systematic Rewievs 2013, Issue 4. Art. No.:CD002822. DOI: 10.1002/14651858.CD2822.pub2. Teemanumeron katsausta ei tulisi kuormittaa viitteillä (max. 30).
- Big data Suomessa. Keskustelualoite 2013
- Big Data muuttaa maailmaa Talouselämä 2013
- 21 polkua kitkattomaan Suomeen
- Data to Intelligence
- fi:Big data
- Big Data -strategia
| Tämä sivu on tiedonmuru. Tämä sivu poikkeaa muusta Opasnetin sisällöstä sen suhteen ettei se ole vapaasti muokattavissa. Käyttäessäsi sivun sisältämää tietoa muualla ole hyvä ja viittaa tähän sivuun näin:
Jouni T. Tuomisto, Antti Poikola: Massadata kansanterveyden edistämisessä. Opasnet 2015. Viitattu 07.05.2024. (, Duodecim)
|
| Tämä sivu on tiedonmuru.
Sivutunniste: Op_fi4995 |
|---|
| Moderaattori:Jouni (katso kaikki)
Sivun edistymistä ei ole arvioitu. Arvostuksen määrää ei ole arvioitu (ks. peer review). |
| Lisää dataa
Tämän Opasnet-sivun julkaisutiedot Jouni T. Tuomisto, Antti Poikola: Massadata kansanterveyden edistämisessä. Opasnet 2015. Viitattu 07.05.2024. (, Duodecim)
|
- ↑ Md. Altaf-Ul-Amin, Farit Mochamad Afendi, Samuel Kuria Kiboi, and Shigehiko Kanaya; Systems Biology in the Context of Big Data and Networks
- ↑ Hamid Bolouri; Modeling genomic regulatory networks with big data
- ↑ Suparna Choudhury, Jennifer R. Fishman , Michelle L. McGowan and Eric T. Juengst; Big data, open science and the brain: lessons learned from genomics. doi: 10.3389/fnhum.2014.00239
- ↑ Christopher G. Chute, MD, DrPH, Mollie Ullman-Cullere, MS, MSE, Grant M. Wood, BS, Simon M. Lin, MD, Min He, PhD, and Jyotishman Pathak, PhD; Some experiences and opportunities for big data in translational research. doi:10.1038/gim.2013.121.
- ↑ J. A. Cook and G. S. Collins; The rise of big clinical databases. DOI: 10.1002/bjs.9723
- ↑ John P Cunningham & Byron M Yu; Dimensionality reduction for large-scale neural recordings. doi:10.1038/nn.3776
- ↑ Lin Dai, Xin Gao, Yan Guo, Jingfa Xiao and Zhang Zhang; Bioinformatics clouds for big data manipulation
- ↑ Steven E. Dilsizian & Eliot L. Siegel; Artificial Intelligence in Medicine and Cardiac Imaging: Harnessing Big Data and Advanced Computing to Provide Personalized Medical Diagnosis and Treatment. DOI 10.1007/s11886-013-0441-8
- ↑ Dong et al. Reverse enGENEering of Regulatory Networks from Big Data: A Roadmap for Biologists. Bioinformatics and Biology Insights 2015:9 61–74 doi: 10.4137/BBI.S12467.
- ↑ Focus on big data. Nature Neuroscience editorial, Volume 17, number 11, November 2014
- ↑ Adam R Ferguson, Jessica L Nielson, Melissa H Cragin, Anita E Bandrowski & Maryann E Martone; Big data from small data: data-sharing in the ‘long tail’ of neuroscience. Nature Neuroscience, Volume 17, number 11, November 2014
- ↑ Alex Gomez-Marin, Joseph J Paton, Adam R Kampff, Rui M Costa & Zachary F Mainen; Big behavioral data: psychology, ethology and the foundations of neuroscience. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3812
- ↑ Casey S. Greene, Jie Tan, Matthew Ung, Jason H. Moore and Chao Cheng; Big Data Bioinformatics. J. Cell. Physiol. 229: 1896–1900, 2014. DOI: 10.1002/jcp.24662
- ↑ Rowland R. Kao, Daniel T. Haydon, Samantha J. Lycett, and Pablo R. Murcia; Omics: Fulfilling the Promise. Supersize me: how whole-genome sequencing and big data are transforming epidemiology
- ↑ Robert R Kitchen, Joel S Rozowsky, Mark B Gerstein & Angus C Nairn; Decoding neuroproteomics: integrating the genome, translatome and functional anatomy. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3829
- ↑ Jeff W Lichtman, Hanspeter Pfister & Nir Shavit; The big data challenges of connectomics. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3829
- ↑ Ian Maze, Li Shen, Bin Zhang, Benjamin A Garcia, Ningyi Shao, Amanda Mitchell, HaoSheng Sun, Schahram Akbarian, C David Allis & Eric J Nestler; Analytical tools and current challenges in the modern era of neuroepigenomics. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3829
- ↑ Ivan Merelli, Horacio Pérez-Sánchez, Sandra Gesing, and Daniele D’Agostino; Managing, Analysing, and Integrating Big Data in Medical Bioinformatics: Open Problems and Future Perspectives, BioMed Research International. Volume 2014, Article ID 134023
- ↑ Russell A Poldrack & Krzysztof J Gorgolewski; Making big data open: data sharing in neuroimaging. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3829
- ↑ Terrence J Sejnowski, Patricia S Churchland, and J Anthony Movshon; Putting big data to good use in neuroscience. Published as: Nat Neurosci. 2014 November ; 17(11): 1440–1441.
- ↑ Terrence J Sejnowski, Patricia S Churchland & J Anthony Movshon; Putting big data to good use in neuroscience. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3829
- ↑ Jaehoon Shin, Guo-li Ming & Hongjun Song; Decoding neural transcriptomes and epigenomes via high-throughput sequencing. Nature Neuroscience, Volume 17, number 11, November 2014, doi:10.1038/nn.3829
- ↑ Weiqi Wang, PhD, Eswar Krishnan, MD, MPH; Big Data and Clinicians: A Review on the State of the Science.
- ↑ Hongyan Wu, Atsuko Yamaguchi; Semantic Web technologies for the big data in life sciences. BioScience Trends. 2014; 8(4):192-201.
- ↑ Changwon Yoo, Luis Ramirez, Juan Liuzzi; Big Data Analysis Using Modern Statistical and Machine Learning Methods in Medicine. http://dx.doi.org/10.5213/inj.2014.18.2.50
- ↑ Dong Zou, Lina Ma, Jun Yu, Zhang Zhang; Biological Databases for Human Research. Genomics Proteomics Bioinformatics 13 (2015) 55–63
- ↑ Viittausvirhe: Virheellinen
<ref>-elementti; viitettädilsizianei löytynyt
