Ero sivun ”Vesiopas” versioiden välillä
pEi muokkausyhteenvetoa |
(→Vastaus: summausvirhe korjattu) |
||
| (36 välissä olevaa versiota 3 käyttäjän tekeminä ei näytetä) | |||
| Rivi 7: | Rivi 7: | ||
[[Luokka:Sisältää R-koodia]] | [[Luokka:Sisältää R-koodia]] | ||
[[Tiedosto:Talousveden_riskinarviointi.png|thumb|500px]] | [[Tiedosto:Talousveden_riskinarviointi.png|thumb|500px]] | ||
'''Vesiopas - Arviointi vedenlaadun terveysvaikutuksista''' käsittelee juomaveden mahdollisia mikrobiologisia terveysriskejä. Mikrobiologisten riskien taustana on raakaveden kontaminoituminen mikrobeilla joista aiheutuu mahdollista terveyshaittaa vesijohtovettä käyttäville ihmisille, sekä juomaveden käsittelyprosessien tehokkuus, joka voi olla riittämätön poistamaan haitalliset mikrobit juomavedestä. Arvioinnilla määritetään, minkä suuruinen mahdollinen terveysriski on tietyillä raakaveden mikrobeilla. Arviointi perustuu matemaattiselle Vesiopas-mallille, joka esitellään alla. | '''Vesiopas - Arviointi vedenlaadun terveysvaikutuksista''' käsittelee juomaveden mahdollisia mikrobiologisia terveysriskejä. Mikrobiologisten riskien taustana on raakaveden kontaminoituminen mikrobeilla joista aiheutuu mahdollista terveyshaittaa vesijohtovettä käyttäville ihmisille, sekä juomaveden käsittelyprosessien tehokkuus, joka voi olla riittämätön poistamaan haitalliset mikrobit juomavedestä. Arvioinnilla määritetään, minkä suuruinen mahdollinen terveysriski on tietyillä raakaveden mikrobeilla. Arviointi perustuu matemaattiselle Vesiopas-mallille, joka esitellään alla. | ||
| Rivi 17: | Rivi 15: | ||
==Vastaus== | ==Vastaus== | ||
[http://fi.opasnet.org/fi-opwiki/index.php?title=Toiminnot:RTools&id=eI6bIUZPZMbplC4u Esimerkkiajo suuren pintavesikuormituksen jälkeen]. | |||
<rcode name="answer" graphics=1 variables=" | <rcode name="answer" graphics=1 variables=" | ||
name: | name:rawclass|description:Raakaveden valmiit luokitukset|type:selection| | ||
options: | options: | ||
' | 'Ground water - clean';Pohjavesi - Puhdas; | ||
' | 'Ground water - surface water contamination';Pohjavesi - Pintavesikuormitus; | ||
' | 'Surface water - low contamination';Pintavesi - Vähäinen kuormitus; | ||
' | 'Surface water - medium contamination';Pintavesi - Keskisuuri kuormitus; | ||
' | 'Surface water - high contamination';Pintavesi - Suuri kuormitus; | ||
' | 'Hand input';Ei valmista luokitusta, määrittelen mikrobipitoisuudet itse| | ||
category:Perustiedot| | category:Perustiedot| | ||
name: | name:rawconsumption|default:0.25|description:Kylmän hanaveden juominen (litraa/pvä)| | ||
name: | name:total_population|default:10000|description:Altistuvan väestön koko| | ||
name: | name:Campylo|description:Kampylobakteerin määrä (mikrobia/l)|default:0| | ||
category: | category:Taudinaiheuttajien määrät raakavedessä (jos ei valmista luokitusta)| | ||
category_conditions:RaakaLuokka;'Käyttäjä'| | category_conditions:RaakaLuokka;'Käyttäjä'| | ||
name: | name:E.coli|description:''E.coli'' O157:H7 määrä (mikrobia/l)|default:0| | ||
name:Rota|description: | name:Rota|description:Rotaviruksen määrä (mikrobia/l)|default:0| | ||
name:Noro|description: | name:Noro|description:Noroviruksen määrä (mikrobia/l)|default:0| | ||
name:Crypto|description: | name:Sapo|description:Sapoviruksen määrä (mikrobia/l)|default:0| | ||
name:Giardia|description: | name:Crypto|description:Cryptosporidiumin määrä (mikrobia/l)|default:0| | ||
name: | name:Giardia|description:Giardian määrä (mikrobia/l)|default:0| | ||
name:treatment|description:Käytössä olevat puhdistusmenetelmät|type:checkbox|options: | |||
'Coagulation and flotation';Saostaminen ja flotaatio; | |||
'Slow sand filtration';Hidas hiekkasuodatus; | |||
'Lime stone filtration';Kalkkikivisuodatus; | |||
'Active carbon filtration';Aktiivihiilisuodatus| | |||
category:Vedenpuhdistus: Puhdistusprosessi ja klooraus| | category:Vedenpuhdistus: Puhdistusprosessi ja klooraus| | ||
name:chlorinedose|default:0|description:Klooriannos (mg/l)| | |||
name:ozonedose|default:0|description:Otsoniannos (mg/l)| | |||
name:uvdose|default:0|description:UV annos (mW/cm^2)| | |||
name:uvtime|default:0|description:UV aika (s) | |||
" | |||
> | |||
# This is code Op_fi2583/answer on page [[Water guide]] | |||
# | |||
library(OpasnetUtils) | library(OpasnetUtils) | ||
library( | library(tidyverse) | ||
# | # Default variables that can be used if no user interface is available | ||
if ( | if(FALSE) { | ||
rawclass <- 'Surface water - high contamination' # ;Pintavesi - Suuri kuormitus; | |||
rawconsumption <- 0.25 # Kylmän hanaveden juominen (litraa/pvä) | |||
total_population <- 10000 # Altistuvan väestön koko | |||
# Campylo <- 0 # Kampylobakteerin määrä (mikrobia/l) | |||
# category:Taudinaiheuttajien määrät raakavedessä (jos ei valmista luokitusta)| | |||
# category_conditions:RaakaLuokka;'Käyttäjä'| | |||
# name:E.coli|description:''E.coli'' O157:H7 määrä (mikrobia/l)|default:0| | |||
# name:Rota|description:Rotaviruksen määrä (mikrobia/l)|default:0| | |||
# name:Noro|description:Noroviruksen määrä (mikrobia/l)|default:0| | |||
# name:Sapo|description:Sapoviruksen määrä (mikrobia/l)|default:0| | |||
# name:Crypto|description:Cryptosporidiumin määrä (mikrobia/l)|default:0| | |||
# name:Giardia|description:Giardian määrä (mikrobia/l)|default:0| | |||
treatment <- NULL # Käytössä olevat puhdistusmenetelmät|type:checkbox|options: | |||
# 'Coagulation and flotation';Saostaminen ja flotaatio; | |||
# 'Slow sand filtration';Hidas hiekkasuodatus; | |||
# 'Lime stone filtration';Kalkkikivisuodatus; | |||
# 'Active carbon filtration';Aktiivihiilisuodatus| | |||
# category:Vedenpuhdistus: Puhdistusprosessi ja klooraus| | |||
chlorinedose <- 0 # Klooriannos (mg/l)| | |||
ozonedose <- 0 # Otsoniannos (mg/l)| | |||
uvdose <- 0 # |description:UV annos (mW/cm^2)| | |||
uvtime <- 0 # |description:UV aika (s) | |||
} | } | ||
objects.latest(" | objects.latest("Op_en6177", code_name="waterguide") # [[Water guide]] fetch the whole model | ||
# Create ovariables from user input data | |||
RawClass <- Ovariable("RawClass", data=data.frame(RawWaterClass = rawclass, Result=1)) | |||
Treatment <- Ovariable("Treatment", data=data.frame(TreatmentMethod=c(treatment, "None"), Result=1)) | |||
ChlorineDose <- Ovariable("ChlorineDose", data=data.frame(Result=chlorinedose)) | |||
UVdose <- Ovariable("UVdose", data=data.frame(Result=uvdose)) | |||
UVtime <- Ovariable("UVtime", data=data.frame(Result=uvtime)) | |||
OzoneDose <- Ovariable("OzoneDose", data=data.frame(Result=ozonedose)) | |||
} | RawConsumption <- Ovariable("RawConsumption", data=data.frame(Result=rawconsumption)) | ||
# divide the given population size to different age groups based on age distribution of all of Finland | |||
population <- Ovariable("population", | |||
dependencies=data.frame( | |||
Name=c("total_population") | |||
), | |||
formula=function (...) { | |||
population2 <- Ovariable( | |||
"population2", | |||
ddata="Op_en2949", subset="Population" | |||
) | |||
population2 <- EvalOutput(population2) | |||
population <- population2/oapply(population2, cols = "Age", FUN=sum) * total_population | |||
return(population) | |||
} | |||
) | |||
# | # if the raw concentrations were chosen to be given by hand, put them into an ovariable | ||
# | # if some raw water class was chosen, this isn't done, and instead the data for microbe concentrations for that class is used | ||
if (rawclass == "Hand input") { | |||
RawConcentration <- Ovariable( | |||
"RawConcentration", | |||
data=data.frame( | |||
Pathogen=c( | |||
"campylobacter", | |||
"E.coli O157:H7", | |||
"rotavirus", | |||
"norovirus", | |||
"sapovirus", | |||
"cryptosporidium", | |||
"giardia"), | |||
Result=c( | |||
Campylo, | |||
E.coli, | |||
Rota, | |||
Noro, | |||
Sapo, | |||
Crypto, | |||
Giardia) | |||
) | |||
) | |||
} | } | ||
BoDattr <- unkeep(EvalOutput(BoDattr, verbose=FALSE, forceEval=TRUE),sources=TRUE, prevresults = TRUE) | |||
BoDattr$Age <- factor(BoDattr$Age, levels=c("0-4","5-9","10-14","15-64","65-79","80+")) | |||
# calculate the number of cases | |||
cases <- BoDattr/case_burden | |||
cat("Raakaveden patogeenipitoisuudet, microbes/l\n") | |||
oprint(summary(RawConcentration)) | |||
cat("Altistuminen, juomaveden mukana nieltyjen mikrobien määrä päivittäin\n") | |||
oprint(summary(exposure)) | |||
cat("Tautitaakka, DALY/päivä (DALY = disability adjusted life years) \n") | |||
oprint(summary(oapply(BoDattr,NULL,sum,"Age"), marginals=c("Response","Exposure_agent"))) | |||
cat("Juomaveden aiheuttamat tautitapaukset päivässä \n") | |||
oprint(summary(oapply(cases,NULL,sum,"Age"), marginals=c("Response", "Exposure_agent"))) | |||
label <- oapply(BoDattr, c("Exposure_agent", "Iter"), sum) | |||
label <- oapply(unkeep(label, sources=TRUE), NULL, mean, "Iter")@output$BoDattrResult | |||
ggplot( | |||
oapply(unkeep(BoDattr, sources=TRUE), NULL, mean, "Iter")@output, | |||
aes(x=Exposure_agent, weight=BoDattrResult, fill=Response)) + | |||
geom_bar() + | |||
coord_flip() + | |||
theme_gray(base_size=24)+ | |||
labs( | |||
title="Juomaveden mikrobien aiheuttama tautitaakka", | |||
subtitle=paste(format(sum(result(population)), scientific = F), "hengen populaatiossa"), | |||
y = "Tautitaakka (DALY/päivä)", | |||
x = "Taudinaiheuttaja") + | |||
annotate("text", x = 1:length(label), y = label, label = signif(label,2), size = 6) | |||
ggplot( | |||
oapply(unkeep(BoDattr, sources=TRUE), NULL, mean, "Iter")@output, | |||
aes(x=Age, weight=BoDattrResult, fill=Response)) + | |||
geom_bar() + | |||
coord_flip() + | |||
theme_gray(base_size=24)+ | |||
labs( | |||
title="Juomaveden mikrobien aiheuttama tautitaakka", | |||
subtitle=paste(format(sum(result(population)), scientific = F), "hengen populaatiossa"), | |||
y = "Tautitaakka (DALY/päivä)", | |||
x = "Taudinaiheuttaja") | |||
label2 <- oapply(cases, c("Exposure_agent", "Iter"), sum) | |||
label2 <- oapply(unkeep(label2, sources=TRUE), NULL, mean, "Iter")@output$Result | |||
ggplot( | |||
oapply(unkeep(cases, sources=TRUE), NULL, mean, "Iter")@output, | |||
aes(x=Exposure_agent, weight=Result, fill=Response)) + | |||
geom_bar() + | |||
coord_flip() + | |||
theme_gray(base_size=24)+ | |||
labs( | |||
title="Juomaveden mikrobeiden aiheuttamat tautitapaukset", | |||
subtitle=paste(format(total_population, scientific = F), "hengen populaatiossa"), | |||
y = "Tautitapaukset", | |||
x = "Taudinaiheuttaja") + | |||
annotate("text", x = 1:length(label2), y = label2, label = signif(label2,2), size = 6) | |||
# cases as percent of beachgoers | |||
cases_percent <- cases*100/total_population | |||
label3 <- oapply(cases_percent, c("Exposure_agent", "Iter"), sum) | |||
label3 <- oapply(unkeep(label3, sources=TRUE), NULL, mean, "Iter")@output$Result | |||
ggplot( | |||
oapply(unkeep(cases_percent, sources=TRUE), NULL, mean, "Iter")@output, | |||
aes(x=Exposure_agent, weight=Result, fill=Response)) + | |||
geom_bar() + | |||
coord_flip() + | |||
theme_gray(base_size=24)+ | |||
labs( | |||
title="Osuus väestöstä, joka sairastuu juomavedestä päivittäin", | |||
y = "Osuus väestöstä (%)", | |||
x = "Taudiniheuttaja") + | |||
annotate("text", x = 1:length(label3), y = label3, label = signif(label3,2), size = 6) | |||
cat(paste("Suomessa juomavedestä johtuvia tautitapauksia tulisi olla keskimäärin vähemmän kuin 0.01% populaatiosta vuosittain, tai 1 tapaus 10 000:ä henkeä kohti. WHO:n ohjeet antavat tavoitetasoksi vähemmän kuin 10^-6 DALYa henkeä kohti vuodessa, eli 0.01 DALYa 10 000:ä henkeä kohti. Nämä kuvaajat näyttävät tautitaakat ja tautitapausten määrän jokainen patogeeni auheuttaa päivittäin", format(sum(result(population)), scientific = F), "hengen populaatiossa, kun kylmää hanavettä juodaan keskimäärin", rawconsumption, "litraa/hlö/päivä. \n")) | |||
</rcode> | </rcode> | ||
Tulossivu avautuu uudelle välilehdelle painamalla aja koodi -laatikkoa. Tulokset | Tulossivu avautuu uudelle välilehdelle painamalla aja koodi -laatikkoa. Tulokset ilmestyvät näkyviin tulossivulle, kunhan malli on saanut ajettua. Tähän menee noin 45 sekuntia. | ||
=== Käyttöohjeet Vesiopas-mallille === | === Käyttöohjeet Vesiopas-mallille === | ||
| Rivi 517: | Rivi 236: | ||
Valitse 'Määrittelen mikrobipitoisuudet itse'. Kirjoita patogeenien pitoisuudet niiden laatikoihin. | Valitse 'Määrittelen mikrobipitoisuudet itse'. Kirjoita patogeenien pitoisuudet niiden laatikoihin. | ||
'''2. Ilmoita juomaveden kulutus''' vuorokautta kohden litroina päivässä henkeä kohti (oletus | '''2. Ilmoita juomaveden kulutus''' vuorokautta kohden litroina päivässä henkeä kohti (oletus 0,796 l/pvä). | ||
'''3. Ilmoita kohdealueen väestön lukumäärä.''' (oletus 100000) | '''3. Ilmoita kohdealueen väestön lukumäärä.''' (oletus 100000) | ||
| Rivi 523: | Rivi 242: | ||
'''4. Valitse puhdistusprosessit.''' Huom! Voit valita monta vaihtoehtoa | '''4. Valitse puhdistusprosessit.''' Huom! Voit valita monta vaihtoehtoa | ||
'''5. Valitse onko klooraus käytössä vai ei''' syöttämällä käytetty klooriannos (mg/l). Mikäli kloorausta ei ole, annokseksi tulee 0 mg/l (oletus | '''5. Valitse onko klooraus käytössä vai ei''' syöttämällä käytetty klooriannos (mg/l). Mikäli kloorausta ei ole, annokseksi tulee 0 mg/l (oletus 0 mg/l). Malli laskee kloorin tuottaman puhdistuman vain laitoksissa tapahtuvalle puhdistumiselle, ja jättää jäännekloorin vaikutuksen veden siirryttyä putkistoon huomiotta. | ||
'''6. Tuloksen saa näkyviin uudelle sivulle painamalla nappia 'Aja koodi'.''' | '''6. Tuloksen saa näkyviin uudelle sivulle painamalla nappia 'Aja koodi'.''' Tämä avaa uuden välilehden, jolle mallin tulokset ilmestyvät sen ajettua. Ajamisessa kestää noin 45 sekuntia. | ||
}} | }} | ||
| Rivi 563: | Rivi 278: | ||
=== Riippuvuudet === | === Riippuvuudet === | ||
{| {{prettytable}} | |||
|+ '''Sivut, joilta mallin käyttämä data ja koodit löytyvät | |||
! Sivu !! Mitä dataa tai koodia sivulla on !! Mitä sillä tehdään !! Muita huomioita | |||
|---- | |||
| Op_en2949 [[:op_en:Population of Finland|Population of Finland]] || Suomalaisten ikäjakauman data || Ikäjakaumaa käytetään, koska herkkyys taudeille ja niiden vakavuus vaihtelee iän myötä. Tautitapaukset ja -taakat lasektaan eri ikäryhmille erikseen ja lasketaan yhteen. || | |||
|---- | |||
| Op_en7956 [[:op_en:Drinking water chlorination efficiency|Drinking water chlorination efficiency]] || Kloorauksen tuottama puhdistuma eri kloorimäärillä ja vaikutusajoilla || Tämän sivun koodin ja datan sekä käyttäjän antaman klooriannoksen avulla lasketaan tapauskohtainen kloorin tuottama puhdistuma. Lasketaan yhteen muiden defintiointi- ja puhdistusmenetelmien tuottaman puhdistuman kanssa kokonaispuhdistuman selvittämiseksi. || Malli ottaa huomioon vain vedenpuhdistamoissa tapahtuvan kloorin tuottaman puhdistuman. Jäännekloorin mahdollinen puhdistava vaikutus putkistoissa on jätetty huomiotta. | |||
|---- | |||
| Op_en7955 [[:op_en:Drinking water disinfection efficiency|Drinking water disinfection efficiency]] || Otsonoinnin ja UV:n tuottamat puhdistumat || Mikäli käyttäjä on valinnut jomman kumman tai molemmat desinfiointimenetelmät, niiden tuottama puhdistuma lasketaan yhteen kloorauksen ja muiden puhdistusmenetelmien tuottaman puhdistuman kanssa kokonaispuhdistuman selvittämiseksi. || Tällä hetkellä veden ominaisuuksia, kuten kirkkautta, ei oteta huomioon desinfiointimeletelmien tehossa. Molempien menetelmien annos on myös vakio. | |||
|---- | |||
| Op_en7954; [[:op_en:Drinking water treatment efficiency|Drinking water treatment efficiency]] || Eri puhdistusmenetelmien tuottamat puhdistukset. Nämä menetelmät ovat perinteinen puhdistus, hyvin toimva puhdistus, tehostettu puhdistus, hidas hiekkasuodatus, kalkkikivisuodatus ja aktiivihiilisuodatus. || Mikäli käyttäjä on valinnut jonkin tai useamman puhdistusmenetelmän, niiden tuottama puhdistuma lasketaan yhteen kloorauksen ja muiden desinfiointimenetelmien tuottaman puhdistuman kanssa kokonaispuhdistuman selvittämiseksi. || | |||
|---- | |||
| Op_en7953 [[:op_en:Pathogen concentrations in raw water|Pathogen concentrations in raw water]] || Eri raakavesiluokkien sisältämät taudinaiheuttajamäärät || Mikäli käyttäjä on valinnut jonkin raakavesiluokan, valitun luokan taudinaiheuttajamäärien ja kokonaispuhdistuman perusteella lasketaan juomavedessä olevien taudinaiheuttajien määrä. Mikäli käyttäjä on syöttänyt mikrobipitooisuudet itse, tämän sivun dataa ei käytetä, vaan syötettyjä mikrobipitoisuuksia käytetään sen sijasta. || | |||
|---- | |||
| Op_en7947 [[:op_en:Case burden of waterborne microbes|Case burden of waterborne microbes]] || Eri taudinaiheuttajien tautitaakat, eli kuinka vakavia ja pitkään kestäviä eri taudit ovat. || Tätä dataa käytetään taudinaiheuttajien tuottaman terveyshaitan laskemisessa. || | |||
|---- | |||
| Op_en7957 [[:op_en:Health impacts of waterborne microbes|Health impacts of waterborne microbes]] || Ei sisällä dataa, vain koodia || Sisältää koodit, jotka laskevat käyttäjän antaman juodun juomaveden määrän, kokonaispuhdistuman ja raakaveden taudinaiheuttajamäärien perusteella yksittäisen ihmisen altistumisen taudinaiheuttajille eli sen, kuinka monta kutakin taudinaiheuttajaa keskivertoihminen nielee päivän aikana juomavetensä mukana. || | |||
|---- | |||
| Op_en7948 [[:op_en:ERF of waterborne microbes|ERF of waterborne microbes]] || Kuvaa annos-vaste-funktiot || Funktiot kertovat, kuinka todennäköisesti yksilö sairastuu eri tauteihin eri määrillä altistumista. Tämän avulla lasketaan sairastuneiden määrää. || | |||
|---- | |||
| Op_en2261 [[:op_en:HIA|HIA]] || Koodi, joka kokoaa yhteen kaikki edelliset || Sivulla oleva koodi laskee altistumisen, annos-vasteiden ja tapauskohtaisten tautitaakkojen avulla, kuinka suuri juomaveden kokonaistautitaakka on. Tästä voidaan laskea myös, kuinka paljon ihmisiä tulee todennäköisesti sairaaksi juomavedestä. Nämä ovat mallin lopulliset tulokset. || | |||
|} | |||
== Katso myös == | == Katso myös == | ||
{{metodi|moderator = Päivi Meriläinen}} | {{metodi|moderator = Päivi Meriläinen}} | ||
* [https://www.vesi.fi/ Ajantasaista vesitietoa vesi.fi] | |||
*[[Juomaveden patogeenit]] | *[[Juomaveden patogeenit]] | ||
*[[Juomaveden patogeenien terveysvaikutukset]] | *[[Juomaveden patogeenien terveysvaikutukset]] | ||
| Rivi 586: | Rivi 313: | ||
* [[Mikrobien määrä juomavedessä]] | * [[Mikrobien määrä juomavedessä]] | ||
* [[Mikrobien määrä pohjavedessä]] | * [[Mikrobien määrä pohjavedessä]] | ||
*[[Uimavesiopas]] | |||
* WHO: [https://apps.who.int/iris/bitstream/handle/10665/246195/9789241565370-eng.pdf?sequence=1 Quantitative Microbial Risk Assessment: Application for Water Safety Management] | |||
* [http://fi.opasnet.org/fi-opwiki/index.php?title=Vesiopas&oldid=35905 Vanha, arkistoitu versio mallista] | |||
*http://qmrawiki.org/ | |||
*https://www.watershare.eu/tool/qmra-treatment-calculator/ | |||
*http://qmrawiki.canr.msu.edu/index.php/Quantitative_Microbial_Risk_Assessment_(QMRA)_Wiki | |||
*https://www.who.int/water_sanitation_health/publications/qmra/en/ | |||
*https://www.rivm.nl/en/who-collaborating-centre-risk-assessment-of-pathogens-in-food-and-water/tools/qmraspot | |||
; Turhia? | ; Turhia? | ||
Nykyinen versio 10. toukokuuta 2021 kello 17.55
Vesiopas - Arviointi vedenlaadun terveysvaikutuksista käsittelee juomaveden mahdollisia mikrobiologisia terveysriskejä. Mikrobiologisten riskien taustana on raakaveden kontaminoituminen mikrobeilla joista aiheutuu mahdollista terveyshaittaa vesijohtovettä käyttäville ihmisille, sekä juomaveden käsittelyprosessien tehokkuus, joka voi olla riittämätön poistamaan haitalliset mikrobit juomavedestä. Arvioinnilla määritetään, minkä suuruinen mahdollinen terveysriski on tietyillä raakaveden mikrobeilla. Arviointi perustuu matemaattiselle Vesiopas-mallille, joka esitellään alla.
Kysymys
Miten arvioidaan juomaveden mikrobiologisia riskejä sekä niiden aiheuttamia terveysvaikutuksia? Erilaisten vedenpuhdistuslaitosten tietoja pitää voida syöttää arviointiin lähtötiedoiksi.
Vastaus
Esimerkkiajo suuren pintavesikuormituksen jälkeen.
Tulossivu avautuu uudelle välilehdelle painamalla aja koodi -laatikkoa. Tulokset ilmestyvät näkyviin tulossivulle, kunhan malli on saanut ajettua. Tähän menee noin 45 sekuntia.
Käyttöohjeet Vesiopas-mallille
1. Valitse raakaveden luokitus
- Pohjavesi - Puhdas: puhdasta pohjavettä
- Pohjavesi - Pintavesikuormitus: esim. rantaimeytymistä
- Pintavesi - Vähäinen kuormitus: suhteellisen puhdasta pintavettä
- Pintavesi - Keskisuuri kuormitus: esim. pienehkö jätevesikuormitus
- Pintavesi - Suuri kuormitus: esim. jätevesikuormitus
TAI
Valitse 'Määrittelen mikrobipitoisuudet itse'. Kirjoita patogeenien pitoisuudet niiden laatikoihin.
2. Ilmoita juomaveden kulutus vuorokautta kohden litroina päivässä henkeä kohti (oletus 0,796 l/pvä).
3. Ilmoita kohdealueen väestön lukumäärä. (oletus 100000)
4. Valitse puhdistusprosessit. Huom! Voit valita monta vaihtoehtoa
5. Valitse onko klooraus käytössä vai ei syöttämällä käytetty klooriannos (mg/l). Mikäli kloorausta ei ole, annokseksi tulee 0 mg/l (oletus 0 mg/l). Malli laskee kloorin tuottaman puhdistuman vain laitoksissa tapahtuvalle puhdistumiselle, ja jättää jäännekloorin vaikutuksen veden siirryttyä putkistoon huomiotta.
6. Tuloksen saa näkyviin uudelle sivulle painamalla nappia 'Aja koodi'. Tämä avaa uuden välilehden, jolle mallin tulokset ilmestyvät sen ajettua. Ajamisessa kestää noin 45 sekuntia.
Perustelut
- Malli on käännetty Analyticasta R:lle. Alkuperäinen malli löytyy täältä: Tiedosto:Vesiopas.ANA. Mikäli käyttäjä ei itse anna patogeenikohtaisia pitoisuuksia, käytetään sivulla Raakaveden patogeenipitoisuudet olevia arvoja.
Talousveden laadulle on asetettu terveysperusteisia laatuvaatimuksia ja -suosituksia. Laatua koskevasta lainsäädännästä vastaa Sosiaali- ja terveysministeriö ja laadun valvonnasta kuntien terveydensuojeluviranomaiset. Hyvän veden laadun ylläpitäminen ja kehittäminen edellyttävät korkealaatuista tutkimusta ja yhteistyötä eri tahojen kanssa. Pohjavesiä tai tekopohjavesiä ei yleensä desinfioida. Raakaveden sisältämät kemikaalit, mikrobit tai talousveden valmistuksessa muodostuvat desinfioinnin sivutuotteet voivat aiheuttaa terveysriskejä talousveden käyttäjille. Vesilaitokselta lähtevä vesi saattaa viipyä pitkiäkin aikoja verkostossa. Mikäli olosuhteet ovat mikrobikasvustolle suotuisat, vesi voi muuttua verkostossa ja kiinteistöjen vesijärjestelmissä huonolaatuiseksi ja jopa terveydelle haitalliseksi.
Talousvesi tuotetaan joko pohja- tai pintavedestä. Näillä on erilaiset ongelmat. Pintaveden käyttö vaatii aina varsinaisen vesilaitoksen perustamista ja siihen liittyvää tietoa, taitoa ja tekniikkaa sekä koulutettua valvontahenkilöstöä. Pohjavesi taas ei vaadi kovin monimutkaista käsittelyä, joten varsinaista vesilaitoshenkilökuntaa tarvitaan varsin vähän ja valvontakin on vähäisempää. Lisäksi meillä on nykyään useita vesilaitoksia, jotka valmistavat tekopohjavettä. Ominaisuuksiltaan tekopohjavedet saattavat olla hyvien pohjavesien kaltaisia, mutta usein vain keskitasoisen pintaveden veroisia.
Pohjaveden käsittelyyn kuuluu tavallisesti veden pH:n ja kovuuden nosto. Tähän voidaan käyttää erilaisia tekniikoita, joista pH:n säädössä kalsiumhydroksidin ja hiilidioksidin käyttö lienee yleisimpiä. Toisinaan tarvitaan lisäksi raudan tai mangaanin saostaminen vahvoilla hapettimilla kuten permanganaatilla tai kloorilla. Lisäksi heikkolaatuiset pohjavedet desinfioidaan.
Jotta pintavedestä saataisiin hyvää juomavettä, sitä joudutaan käsittelemään varsin rankasti. Vaikeinta on saada pintaveden humusainekset saostettua. Tähän käytetään erilaisten saostuskemikaalien lisäksi mm. pH:n säätöä ja humusaineksen pilkkomista voimakkailla hapettimilla kuten otsonilla. Lisäksi saostuskemikaalien (esim. alumiinin ja raudan suolat) jäämät on poistettava ja veden kovuutta nostettava niin, ettei putkisto aikojen kuluessa haperru. Väestö on myös suojattava pintavesissä aina piileviltä mikrobeilta, joten vesi täytyy desinfioida tautia aiheuttavien mikrobien, patogeenien, poistamiseksi.
Veden laatuun vaikuttavat myös verkoston kunto ja veden verkostossa viipymä aika. Jos verkostoon on kertynyt runsaasti saostumia, nämä sakat sisältävät erilaisten epäorgaanisten yhdisteiden lisäksi valtavasti bakteerimassaa. Verkoston saostumien ominaisuuksia ja niiden vaikutusta veden laatuun on Suomessa alettu tutkia vasta viime aikoina.
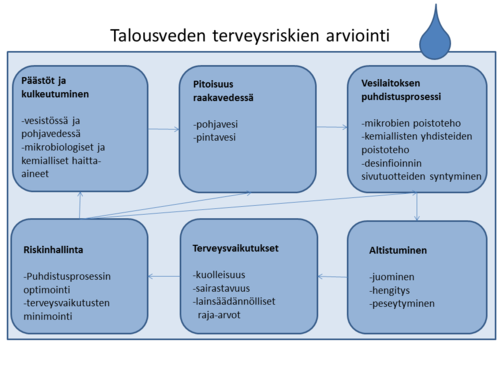
Riskinarvioinnin tarkoituksena on tuottaa tietoa riskin todellisesta suuruudesta päätöksentekoa varten. Riskinarviointi on tieteellinen prosessi, jossa asiantuntijoilla on keskeinen rooli. Koska tutkittavia riskejä monenlaisia, riskinarviointiprosessit voivat olla hyvin erilaisia. Ympäristöterveyden alalla riskinarvioinnin katsotaan tavallisesti koostuvan neljästä vaiheesta:
- Vaaran tunnistaminen (aiheuttaako tutkittava tekijä vaaraa ihmiselle?)
- Annosvasteen arviointi (mikä on annoksen ja vaikutuksen suhde?)
- Altistumisen arviointi (kuinka paljon ihmiset altistuvat?)
- Riskin karakterisointi (kuinka suuri on terveysriski?)[1]
Kaikkien vesilaitosten tulisi ajoittain tehdä talousvedelle riskinarviointi. Siinä selvitetään toimenpiteet eri kriisitilanteita varten ja laaditaan suunnitelmat eri ongelmatilanteita ja mahdollisia vahinkoja varten. Erilaiset vesilaitokset ja esimerkiksi lomakeskukset tarvitsevat erilaiset suunnitelmat ja toteutukset.
Riippuvuudet
| Sivu | Mitä dataa tai koodia sivulla on | Mitä sillä tehdään | Muita huomioita |
|---|---|---|---|
| Op_en2949 Population of Finland | Suomalaisten ikäjakauman data | Ikäjakaumaa käytetään, koska herkkyys taudeille ja niiden vakavuus vaihtelee iän myötä. Tautitapaukset ja -taakat lasektaan eri ikäryhmille erikseen ja lasketaan yhteen. | |
| Op_en7956 Drinking water chlorination efficiency | Kloorauksen tuottama puhdistuma eri kloorimäärillä ja vaikutusajoilla | Tämän sivun koodin ja datan sekä käyttäjän antaman klooriannoksen avulla lasketaan tapauskohtainen kloorin tuottama puhdistuma. Lasketaan yhteen muiden defintiointi- ja puhdistusmenetelmien tuottaman puhdistuman kanssa kokonaispuhdistuman selvittämiseksi. | Malli ottaa huomioon vain vedenpuhdistamoissa tapahtuvan kloorin tuottaman puhdistuman. Jäännekloorin mahdollinen puhdistava vaikutus putkistoissa on jätetty huomiotta. |
| Op_en7955 Drinking water disinfection efficiency | Otsonoinnin ja UV:n tuottamat puhdistumat | Mikäli käyttäjä on valinnut jomman kumman tai molemmat desinfiointimenetelmät, niiden tuottama puhdistuma lasketaan yhteen kloorauksen ja muiden puhdistusmenetelmien tuottaman puhdistuman kanssa kokonaispuhdistuman selvittämiseksi. | Tällä hetkellä veden ominaisuuksia, kuten kirkkautta, ei oteta huomioon desinfiointimeletelmien tehossa. Molempien menetelmien annos on myös vakio. |
| Op_en7954; Drinking water treatment efficiency | Eri puhdistusmenetelmien tuottamat puhdistukset. Nämä menetelmät ovat perinteinen puhdistus, hyvin toimva puhdistus, tehostettu puhdistus, hidas hiekkasuodatus, kalkkikivisuodatus ja aktiivihiilisuodatus. | Mikäli käyttäjä on valinnut jonkin tai useamman puhdistusmenetelmän, niiden tuottama puhdistuma lasketaan yhteen kloorauksen ja muiden desinfiointimenetelmien tuottaman puhdistuman kanssa kokonaispuhdistuman selvittämiseksi. | |
| Op_en7953 Pathogen concentrations in raw water | Eri raakavesiluokkien sisältämät taudinaiheuttajamäärät | Mikäli käyttäjä on valinnut jonkin raakavesiluokan, valitun luokan taudinaiheuttajamäärien ja kokonaispuhdistuman perusteella lasketaan juomavedessä olevien taudinaiheuttajien määrä. Mikäli käyttäjä on syöttänyt mikrobipitooisuudet itse, tämän sivun dataa ei käytetä, vaan syötettyjä mikrobipitoisuuksia käytetään sen sijasta. | |
| Op_en7947 Case burden of waterborne microbes | Eri taudinaiheuttajien tautitaakat, eli kuinka vakavia ja pitkään kestäviä eri taudit ovat. | Tätä dataa käytetään taudinaiheuttajien tuottaman terveyshaitan laskemisessa. | |
| Op_en7957 Health impacts of waterborne microbes | Ei sisällä dataa, vain koodia | Sisältää koodit, jotka laskevat käyttäjän antaman juodun juomaveden määrän, kokonaispuhdistuman ja raakaveden taudinaiheuttajamäärien perusteella yksittäisen ihmisen altistumisen taudinaiheuttajille eli sen, kuinka monta kutakin taudinaiheuttajaa keskivertoihminen nielee päivän aikana juomavetensä mukana. | |
| Op_en7948 ERF of waterborne microbes | Kuvaa annos-vaste-funktiot | Funktiot kertovat, kuinka todennäköisesti yksilö sairastuu eri tauteihin eri määrillä altistumista. Tämän avulla lasketaan sairastuneiden määrää. | |
| Op_en2261 HIA | Koodi, joka kokoaa yhteen kaikki edelliset | Sivulla oleva koodi laskee altistumisen, annos-vasteiden ja tapauskohtaisten tautitaakkojen avulla, kuinka suuri juomaveden kokonaistautitaakka on. Tästä voidaan laskea myös, kuinka paljon ihmisiä tulee todennäköisesti sairaaksi juomavedestä. Nämä ovat mallin lopulliset tulokset. |
Katso myös
| Moderaattori:Päivi Meriläinen (katso kaikki)
Sivun edistymistä ei ole arvioitu. Arvostuksen määrää ei ole arvioitu (ks. peer review). |
| Lisää dataa
|
- Arviointi pohjavesilaitoksen mikrobiologisista riskeistä
- Arviointi juomaveden kloorifenolien terveysvaikutuksista
- heande:Kuovesi
- Mikrobien määrä juomavedessä
- Mikrobien määrä pohjavedessä
- Uimavesiopas
- WHO: Quantitative Microbial Risk Assessment: Application for Water Safety Management
- http://qmrawiki.org/
- https://www.watershare.eu/tool/qmra-treatment-calculator/
- http://qmrawiki.canr.msu.edu/index.php/Quantitative_Microbial_Risk_Assessment_(QMRA)_Wiki
- https://www.who.int/water_sanitation_health/publications/qmra/en/
- https://www.rivm.nl/en/who-collaborating-centre-risk-assessment-of-pathogens-in-food-and-water/tools/qmraspot
- Turhia?
- Infektion vuosittainen todennäköisyys
- Vatsatautien tapaukset
- Patogeenien DALYt vatsataudille Yhdistettävä sivuun op_en:DALY?
Paikkakuntakohtaiset tiedot
- Op_fi2603 Arviointi juomaveden laadun terveysvaikutuksista/Kuopio (vedenpuhdistamon tietoja)
- heande:Polaris:Raakaveden patogeenipitoisuudet/Lahti
- heande:Polaris:Raakaveden patogeenipitoisuudet/Mikkeli
- heande:Polaris:Raakaveden patogeenipitoisuudet/Kirkkonummi
- heande:Polaris:Paikkakuntakohtaiset tiedot talousveden käsittelyyn/Lahti
- heande:Polaris:Paikkakuntakohtaiset tiedot talousveden käsittelyyn/Mikkeli
- heande:Polaris:Paikkakuntakohtaiset tiedot talousveden käsittelyyn/Kirkkonummi
- heande:Polaris:Juomaveden desinfiointi/Lahti
- heande:Polaris:Juomaveden desinfiointi/Mikkeli
- heande:Polaris:Juomaveden desinfiointi/Kirkkonummi
- heande:Polaris:Vedenkäsittelyn tehokkuus/Lahti
- heande:Polaris:Vedenkäsittelyn tehokkuus/Mikkeli
- heande:Polaris:Vedenkäsittelyn tehokkuus/Kirkkonummi
Viitteet

|

|