Kotimaisen kalan edistämisohjelma
| [show] |
|---|
| Moderaattori:Jouni (katso kaikki)
Sivun edistymistä ei ole arvioitu. Arvostuksen määrää ei ole arvioitu (ks. peer review). |
| Lisää dataa
|
Sisällys
Kysymys
Mikä olisi kalansyönnin lisääntymisen terveysvaikutus Suomessa?
Oletettu käyttö ja käyttäjät
Tässä arvioinnissa tarkastellaan skenaarioita, joissa suomalaisten kalansyöntiä lisättäisiin kotimaisen kalan edistämisohjelmalla. Tietoja käyttäisivät mm. Luke, MMM ja YM.
Osallistujat
- Jouni Tuomisto, THL
Rajaus
Tilannetta tarkastelleen toisaalta toteutuneeseen 2017 tilanteeseen ja toisaalta skenaarioihin vuodelle 2027, kun on joko lisätty kotimaisen kalan tuotantoa tai tuontia.
Vaihtoehdot
- Kotimaisen kalan käytön lisääminen
- Viljellyn kalan tuonnin lisääminen
Aikataulu
Alustavia arvioita pyritään tuottamaan maaliskuun 2020 aikana.
Vastaus
Tulokset
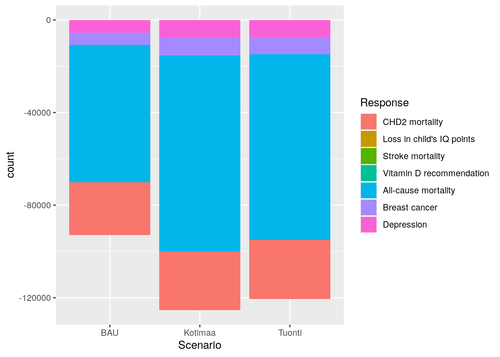
Arviointimallin keskeiset tulokset löytyvät tästä HTML-tiedostosta.
Päätelmät
Kalansyönti vuonna 2017 vähensi tautitaakkaa n. 100 000 haittapainotettua elinvuotta (DALY) Suomessa. Jos kalansyönti lisääntyisi oheisen taulukon mukaisesti, tautitaakka vähenisi edelleen n. 40 000 DALY /a suomalaista kalankulutusta lisäämällä ja n. 35 000 DALY /a kalantuontia lisäämällä.
Perustelut
Data
| Kalaryhmä | BAU 2017 | Kotimainen edistäminen | Tuonnin edistäiminen |
|---|---|---|---|
| Kirjolohi suomalainen | 11 | 45 | 11 |
| Kasvatettu muu suomalainen | 1 | 8 | 3 |
| Silakka | 4 | 18 | 8 |
| Kaupallinen luonnonkala | 6 | 12 | 8 |
| Vapaa-ajan saalis | 20 | 20 | 20 |
| Tuontilohi | 36 | 38 | 67 |
| Tuontikirjolohi | 8 | 8 | 17 |
| Muu tuonti | 28 | 28 | 28 |
Eräs keskeisistä terveysvaikutuksiin vaikuttavista oletuksista on, kuka lisääntyneen kalan söisi. Mahdollisia tapoja ajatella:
- Syöntiä muutetaan nykysyönnin suhteessa.
- Jokainen suomalainen syö vakiomäärän kalaa enemmän.
- Kalansyönnin edistämistä suunnataan tietyille väestöryhmille, ja vaikutukset tulevat siellä (mihin?).
- Vaikutukset jakautuvat satunnaisessa määrin eri yksilöille.
Malliparametrit
Malliparametrit saat näkyviin klikkaamalla.
| [show]Näytä yksityiskohdat | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
Kalankulutus on ilmoitettu fileepainona eikä tuorepainona, eli oletetaan että kaikki tämä tulee syödyksi.
|
Scenario|Action|exposure|Exposure: To child; Age: Age 1|Replace|0.98 (0.17 - 2.9)|Based on RUORI modelling (see code)
Scenario|Action|exposure|Exposure: To eater; Age: Female 18-45|Replace|2.79 (0.56 - 7.08)|Based on RUORI modelling (see code)
Scenario|Action|exposure|Exposure: To eater; Age: Non female 18-45|Replace|11.38 (3.44 - 46.22)|Based on RUORI modelling (see code)
Laskenta
Avoimen arvioinnin mallikoodi on kirjoitettu R:llä ja se löytyy Githubista.
Keskeiset tulosteet
# This is code Op_fi5923/ on page [[Ruori]]
library(OpasnetUtils)
library(ggplot2)
library(thlGraphs)
library(plotly)
openv.setN(10)
objects.latest("Op_fi5923", code_name="model")
# First empty all objects for a fresh start. Otherwise may be problems with CheckDecisions.
oempty(all=TRUE)
InpBoD <- EvalOutput(InpBoD)
InpPAF <- EvalOutput(InpPAF)
utility <- EvalOutput(utility, verbose=TRUE)
# Sample from default and sensitivity scenario about lead threshold.
cat("Elintarvikeperäisen lyijyn vaikutukset herkkyystarkastelussa.\n")
oprint(summary(BoDattr[BoDattr$Exposure_agent=="Lyijy",]))
BoDattr <- CollapseMarginal(BoDattr,"Threshold","sample")
utility <- CollapseMarginal(utility,"Threshold","sample")
levels(BoDattr$Exposure_agent)[levels(BoDattr$Exposure_agent)=="Vihannesvaje"] <- "Kasvisvaje"
#levels(BoDattr$Response)
#[1] "Cancer morbidity" "IQ loss" "Listeriosis" "Liver cancer"
#[5] "Noro infection" "Sperm concentration" "Toxoplasmosis" "Yes or no dental defect"
#[9] "CHD death" "Diet high in sodium" "Diet low in fruits" "Diet low in vegetables"
levels(BoDattr$Response) <- c(
"Syöpä",
"Älykkyysosamäärän lasku",
"Listerioosi",
"Maksasyöpä",
"Noroinfektio",
"Miehen hedelmättömyys",
"Toksoplasmoosi",
"Hammasvaurio",
"Sydäntauti",
"Liika suola",
"Hedelmävaje",
"Kasvisvaje"
)
cat("Elintarvikeperäisiä tautitaakkoja Suomessa arpoen lyijylle oletetun tai matalamman kynnysarvon.\n")
tmp <- summary(oapply(BoDattr[BoDattr$Scenario=="BAU",],NULL,sum,c("Age","Response")))
oprint(data.frame(
Altiste = tmp$Exposure_agent,
Keskiarvo = signif(tmp$mean,2),
"95 luottamusväli" = paste0(signif(tmp$Q0.025,2)," - ", signif(tmp$Q0.975,2)),
Keskihajonta = signif(tmp$sd,2)
)[rev(match(lev, tmp$Exposure_agent)),])
cat("Elintarvikeperäisiä tautitaakkoja Suomessa ikä- ja tautiryhmittäin arpoen lyijylle oletetun tai matalamman kynnysarvon.\n")
tmp <- summary(BoDattr[BoDattr$Scenario=="BAU",])
oprint(data.frame(
Altiste = tmp$Exposure_agent,
Ikä = tmp$Age,
Vaste = tmp$Response,
Keskiarvo = signif(tmp$mean,2),
Mediaani = signif(tmp$median,2),
"95 luottamusväli" = paste0(signif(tmp$Q0.025,2)," - ", signif(tmp$Q0.975,2)),
Keskihajonta = signif(tmp$sd,2)
))
cat("Ruori-skenaarioiden vaikutus tautitaakkaan\n")
tmp <- summary(utility)
oprint(data.frame(
Altiste = tmp$Exposure_agent,
Keskiarvo = signif(tmp$mean,2),
"95 luottamusväli" = paste0(signif(tmp$Q0.025,2)," - ", signif(tmp$Q0.975,2)),
Keskihajonta = signif(tmp$sd,2)
)[rev(match(lev, tmp$Exposure_agent)),])
dodge <- position_dodge(width=0.7)
if(FALSE) {
gg <- ggplot(summary(oapply(BoDattr[BoDattr$Scenario=="BAU",],NULL,sum,"Age")),
aes(x=Exposure_agent, weight=unlist(mean), fill=Response))+geom_bar()+
theme(legend.position = "bottom")+
labs(
title="Elintarvikkeiden tautitaakkoja Suomessa",
subtitle="Haittapainotettua elinvuotta vuodessa (DALY/a)")+
coord_flip()
print(gg)
gg <- ggplot(summary(oapply(BoDattr, NULL, sum,c("Age","Response"))),
aes(x=Exposure_agent, weight=unlist(mean), fill=Scenario))+geom_bar(position="dodge")+
coord_flip(ylim=c(0,70000))+
labs(
title="Elintarvikeperäisiä tautitaakkoja Suomessa",
subtitle="Haittapainotettua elinvuotta vuodessa (DALY/a)")+
geom_errorbar(aes(ymin=unlist(Q0.025),ymax=unlist(Q0.975),group=Scenario),position=dodge, width=0.3)+
geom_text(aes(label=signif(unlist(mean),2), y=unlist(Q0.975)+5000, group=Scenario), position=dodge)
print(gg)
# Utility of actions
gg <- ggplot(summary(utility),aes(x=Exposure_agent, weight=unlist(mean)))+geom_bar(fill="lightblue")+
coord_flip(ylim=c(-9000,0))+
labs(
title="Ruori-skenaarioiden vaikutus tautitaakkaan",
subtitle="Haittapainotettua elinvuotta vuodessa (DALY/a)")+
geom_errorbar(aes(ymin=unlist(Q0.025),ymax=unlist(Q0.975)), width=0.3)+
geom_text(aes(label=signif(unlist(mean),2), y=unlist(Q0.025)-600))
print(gg)
} else {
###### RUN THESE ON OWN COMPUTER WITH thlGraphs PACKAGE
# levels(BoDattr$Exposure_agent)
# [1] "Aflatoksiini" "Dioksiini" "Norovirus" "Toksoplasma"
# [5] "Lyijy" "Listeria" "Tyydyttynyt rasva" "Vihannesvaje"
# [9] "Suola" "Hedelmävaje"
levels(BoDattr$Exposure_agent) <- c(
"Aflatoxin","Dioxin","Noro virus","Toxoplasma", "Lead","Listeria",
"Saturated fat","Lack of vegetables","Sodium","Lack of fruits")
thlBarPlot(summary(oapply(BoDattr[BoDattr$Scenario=="BAU",],NULL,sum,"Age")),
xvar=Exposure_agent, yvar=unlist(mean), groupvar=Response, legend.position="bottom",
colors=thlColors(n=12),
title="Elintarvikkeiden tautitaakkoja Suomessa", subtitle="Haittapainotettua elinvuotta vuodessa (DALY/a)")+coord_flip()
thlBarPlot(summary(oapply(BoDattr, NULL, sum,c("Age","Response"))),xvar=Exposure_agent, yvar=unlist(mean),
groupvar=Scenario,stacked=FALSE, ylimits=c(0,70000),
# title="Elintarvikeperäisiä tautitaakkoja Suomessa",
# subtitle="Haittapainotettua elinvuotta vuodessa (DALY/a)")+
title="Burden of disease of selected food-mediated risk factors in Finland",
subtitle="Disability-adjusted life years per year (DALY/a)")+
coord_flip()+
geom_errorbar(aes(ymin=unlist(Q0.025),ymax=unlist(Q0.975),group=Scenario),position=dodge, width=0.3)+
geom_text(aes(label=signif(unlist(mean),2), y=unlist(Q0.975)+5000, group=Scenario), position=dodge)
ggsave("Ruori Burden of disease.png",width=10,height=6)
# ggsave("Ruori-tautitaakka.png",width=10,height=6)
# Error bars are not used in this plotly because it is unclear what it means in a stacked bar.
p <-plot_ly(
summary(oapply(BoDattr[BoDattr$Scenario=="BAU",], NULL, sum,c("Age"))),
y=~Exposure_agent,
x=~signif(mean,3),
text=~Response,
name=~Response,
type="bar",
orientation = "h"
) %>%
layout(
barmode="stack",
title="Elintarvikeperäisiä tautitaakkoja Suomessa",
xaxis=list(title="Haittapainotettua elinvuotta vuodessa (DALY/a)"),
yaxis=list(title="")
)
# pushIndicatorGraph(p, 117)
# ggsave("Ruori-tautitaakat.png",width=10/1.2,height=6/1.2)
# Utility of actions
thlBarPlot(summary(utility),xvar=Exposure_agent, yvar=unlist(mean), ylimits=c(-9000,0),
title="Ruori-skenaarioiden vaikutus tautitaakkaan",
subtitle="Haittapainotettua elinvuotta vuodessa (DALY/a)")+coord_flip()+
geom_errorbar(aes(ymin=unlist(Q0.025),ymax=unlist(Q0.975)), width=0.3)+
geom_text(aes(label=signif(unlist(mean),2), y=unlist(Q0.025)-600))
# ggsave("Ruori-toimenpideiden vaikutus.png",width=10/1.2,height=6/1.2)
}
|
Arviointimallin alustus
# This is code Op_fi5923/model on page [[Ruori]]
library(OpasnetUtils)
library(ggplot2)
# First empty all objects for a fresh start. Otherwise may be problems with CheckDecisions.
oempty(all=TRUE)
openv.setN(1000)
dat <- opbase.data("Op_fi5923", subset="Malliparametrit")[-1]
dec <- opbase.data("Op_fi5923", subset="Decisions")[-1]
DecisionTableParser(dec)
CTable <- opbase.data("Op_fi5923",subset="CollapseMarginals")
for(i in 1:ncol(CTable)) {CTable[[i]] <- as.character(CTable[[i]])}
CollapseTableParser(CTable)
cat("Laskennassa käytetty data.\n")
oprint(dat)
cat("Tarkastellut päätökset.\n")
oprint(dec)
cat("Aggregoidut marginaalit.\n")
oprint(CTable)
#' prepare adjusts the data table for ovariables. Requires function subgrouping from code Op_en2031/initiate on page [[Exposure-response function]]
#' @param dat data.frame
#' @param type type of data that is used. Must match content in column Type
#' @param drop columns to remove
#' @return data.frame
prepare <- function(dat, type=NULL, drop=NULL) {
out <- dat
if(!is.null(type)) out <- out[out$Type %in% type , ]
if(!is.null(drop)) out <- out[!colnames(out) %in% drop]
return(subgrouping(out))
}
objects.latest("Op_en2031", code_name="subgrouping") # [[Exposure-response function]] subgrouping
population <- Ovariable("population", data = prepare(dat,"population",c("Type","Exposure_agent","Response","Unit")))
exposure <- Ovariable("exposure", data = prepare(dat, "exposure", c("Type","Response")))
frexposed <- Ovariable("frexposed", data=prepare(dat, "frexposed", c("Type","Response","Unit")))
incidence <- Ovariable("incidence", data = prepare(dat,"incidence",c("Type","Exposure_agent","Unit")))
ERFChoice <- Ovariable(
"ERFchoice",
data=data.frame(
Response=c("IQ loss","Liver cancer","Yes or no dental defect","Cancer morbidity","Sperm concenctration"),
Exposure_agent=c("Lead","Aflatoxin","TEQ","TEQ","TEQ"),
Result=1)
)
case_burden <- Ovariable("case_burden", data= prepare(dat,"case burden",c("Type","Exposure_agent","Unit")))
InpPAF <- EvalOutput(Ovariable("InpPAF", data = prepare(dat,"PAF","Type")))
InpBoD <- EvalOutput(Ovariable("InpBoD", data = prepare(dat, "BoD", c("Type","Exposure_agent"))))
objects.latest("Op_en2261",code_name="BoDattr2") # [[Health impact assessment]]
#levels(BoDattr$Exposure_agent)
#[1] "Aflatoxin" "Lead" "TEQ" "Fruits" "Listeria" "Noro virus" "Saturated fat" "Sodium"
#[8] "Toxoplasma gondii" "Vegetables"
BoDattrOrigFormula <- BoDattr@formula
BoDattr@formula <- function(...) {
BoDattr <- BoDattrOrigFormula()
levels(BoDattr$Exposure_agent) <- c("Aflatoksiini","Lyijy", "Dioksiini", "Hedelmävaje","Listeria","Norovirus",
"Tyydyttynyt rasva","Suola", "Toksoplasma","Vihannesvaje")
lev <- oapply(BoDattr[BoDattr$Scenario=="BAU",],"Exposure_agent",sum)
lev <- lev$Exposure_agent[order(result(lev))]
BoDattr$Exposure_agent <- factor(BoDattr$Exposure_agent, levels=lev)
return(BoDattr)
}
utility <- Ovariable(
"utility",
dependencies = data.frame(Name="BoDattr"),
formula = function(...) {
out <- BoDattr * Ovariable(data=data.frame(Scenario=c("Action","BAU"),Result=c(1,-1)))
out <- oapply(out, cols=c("Scenario","Response","Age"),FUN=sum)
return(out)
}
)
utility <- EvalOutput(utility)
lev <- levels(BoDattr$Exposure_agent)
cat("exposure\n")
oprint(summary(exposure,marginals=c("Exposure_agent","Age","Scenario")),digits=7)
oprint(exposure@output[exposure$Iter==1,])
cat("dose\n")
oprint(summary(dose),digits=7)
cat("ERF\n")
oprint(ERF@output[ERF$Iter==1,],digits=7)
cat("incidence\n")
oprint(summary(incidence),digits=7)
cat("frexposed\n")
oprint(summary(frexposed),digits=7)
cat("PAF\n")
oprint(summary(PAF),digits=7)
cat("BoD\n")
oprint(summary(BoD),digits=7)
cat("BoDattr\n")
oprint(summary(BoDattr,marginals=c("Response","Exposure_agent","Scenario","Age")),digits=7)
cat("Vähennyspotentiaali\n")
tmp <- summary(utility, marginals=c("Exposure_agent"))
oprint(tmp[order(unlist(tmp$mean)),])
objects.store(list=setdiff(ls(), "wiki_username"))
cat("Objects", setdiff(ls(), "wiki_username"), "stored.\n")
###################
# Disease burden
# Sample from default and sensitivity scenario about lead threshold.
cat("Elintarvikeperäisen lyijyn vaikutukset herkkyystarkastelussa.\n")
oprint(summary(BoDattr[BoDattr$Exposure_agent=="Lyijy",]))
BoDattr <- CollapseMarginal(BoDattr,"Threshold","sample")
utility <- CollapseMarginal(utility,"Threshold","sample")
cat("Elintarvikeperäisiä tautitaakkoja Suomessa\n")
tmp <- summary(oapply(BoDattr[BoDattr$Scenario=="BAU",],NULL,sum,c("Age","Response")))
oprint(data.frame(
Altiste = tmp$Exposure_agent,
Keskiarvo = signif(tmp$mean,2),
"95 luottamusväli" = paste0(signif(tmp$Q0.025,2)," - ", signif(tmp$Q0.975,2)),
Keskihajonta = signif(tmp$sd,2)
)[rev(match(lev, tmp$Exposure_agent)),])
cat("Ruori-skenaarioiden vaikutus tautitaakkaan\n")
tmp <- summary(utility)
oprint(data.frame(
Altiste = tmp$Exposure_agent,
Keskiarvo = signif(tmp$mean,2),
"95 luottamusväli" = paste0(signif(tmp$Q0.025,2)," - ", signif(tmp$Q0.975,2)),
Keskihajonta = signif(tmp$sd,2)
)[rev(match(lev, tmp$Exposure_agent)),])
dodge <- position_dodge(width=0.7)
ggplot(summary(oapply(BoDattr[BoDattr$Scenario=="BAU",],NULL,sum,"Age")),
aes(x=Exposure_agent, weight=unlist(mean), fill=Response))+geom_bar()+
theme(legend.position = "bottom")+
labs(
title="Elintarvikkeiden tautitaakkoja Suomessa",
subtitle="Haittapainotettua elinvuotta vuodessa (DALY/a)")+
coord_flip()
ggplot(summary(oapply(BoDattr, NULL, sum,c("Age","Response"))),
aes(x=Exposure_agent, weight=unlist(mean), fill=Scenario))+geom_bar(position="dodge")+
coord_flip(ylim=c(0,70000))+
labs(
title="Elintarvikeperäisiä tautitaakkoja Suomessa",
subtitle="Haittapainotettua elinvuotta vuodessa (DALY/a)")+
geom_errorbar(aes(ymin=unlist(Q0.025),ymax=unlist(Q0.975),group=Scenario),position=dodge, width=0.3)+
geom_text(aes(label=signif(unlist(mean),2), y=unlist(Q0.975)+5000, group=Scenario), position=dodge)
# Utility of actions
ggplot(summary(utility),aes(x=Exposure_agent, weight=unlist(mean)))+geom_bar(fill="lightblue")+
coord_flip(ylim=c(-9000,0))+
labs(
title="Ruori-skenaarioiden vaikutus tautitaakkaan",
subtitle="Haittapainotettua elinvuotta vuodessa (DALY/a)")+
geom_errorbar(aes(ymin=unlist(Q0.025),ymax=unlist(Q0.975)), width=0.3)+
geom_text(aes(label=signif(unlist(mean),2), y=unlist(Q0.025)-600))
################ Insight network
gr <- scrape(type="assessment")
objects.latest("Op_en3861", "makeGraph") # [[Insight network]]
gr <- makeGraph(gr)
#export_graph(gr, "ruori.svg")
render_graph(gr)
|